חלק א’: הפקת DNA - מאיפה מתחילים?
בשביל כל בדיקה גנטית, צריך להפיק תחילה DNA. מסתבר שזאת לא משימה טריוויאלית.
מקור הדגימה - למה זה חשוב?
המקור הנפוץ ביותר לדגימות DNA הוא דם, אבל לא תמיד זאת הבחירה המתאימה. המרצה סיפרה על מקרה קליני שנתקלה בו: מטופל הגיע עם בעיה רפואית, ונלקחה ממנו דגימת דם לצורך בדיקת DNA. רק לאחר מכן עלתה המחשבה הקריטית - המטופל קיבל עירוי דם זמן קצר לפני כן. המשמעות היא שהדם שנבדק מכיל גם DNA של התורם, ולא רק של המטופל עצמו, ולכן תוצאת הבדיקה אינה מהימנה.
המסקנה החשובה: תמיד יש לשקול היטב את מקור הדגימה. במטופלים שקיבלו עירויי דם או מוצרי דם, יש לבחור מקור חלופי לדגימת DNA, ולא להסתמך על דם היקפי.
כשרוצים לבדוק גידול
נקודה חשובה שעלתה היא שלא תמיד מעוניינים לבדוק את הגנטיקה של המטופל עצמו, אלא דווקא את הגנטיקה של הגידול. תאים סרטניים צוברים עם הזמן מוטציות סומטיות ייחודיות, שהן אלו שהובילו להתפתחות הגידול ולהתנהגותו. לכן, כאשר מפיקים DNA מדגימת גידול, מתקבל הפרופיל הגנטי של הגידול עצמו - ולא של שאר תאי הגוף של המטופל.
המידע הזה קריטי במיוחד ברפואה מותאמת אישית, משום שהוא מאפשר לזהות מטרות מולקולריות ולקבוע טיפולים ממוקדים שמכוונים למוטציות הספציפיות של הגידול.
איך נראה DNA שהופק?
DNA לאחר הפקה אינו נראה כמו משהו “מעבדתי” קלאסי. בפועל, מדובר בחומר לבן-חלבי, צמיג וסיבי, שלעיתים ניתן לראותו בעין בלתי מזוינת. המרצה תיארה זאת בצורה ציורית כדי להמחיש את המרקם: חומר סמיך ועשיר ב-DNA, בדומה לחומר ביולוגי שמכיל תאים מפורקים וריכוז גבוה של חומצות גרעין.
ההשוואה נועדה להדגיש נקודה חשובה: כשתאים מתפרקים ומשתחרר מהם DNA בכמות גדולה, מתקבל חומר בעל מרקם צמיגי וסיבי (״נזלת״), שהוא מאפיין אופייני של DNA לאחר הפקה מוצלחת.
חלק ב׳: PCR - הבדיקה הבסיסית ביותר בגנטיקה
רקע היסטורי
PCR (Polymerase Chain Reaction) פותחה בשנות ה־90 וזיכתה את מפתחיה בפרס נובל. הודגש בהרצאה שזאת הבדיקה הבסיסית ביותר בגנטיקה — ובלעדיה מרבית הגנטיקה המעבדתית המודרנית לא הייתה מתאפשרת.
העיקרון: הגברה של מקטע מוגדר
הגנום האנושי כולל מיליארדי בסיסים, אך לעיתים נדרש ניתוח של אזור קטן ומדויק בלבד — למשל אקסון מסוים בגן מסוים. PCR מאפשרת הגברה ממוקדת של אותו מקטע, באמצעות יצירת מיליוני עותקים ממנו לצורך עבודה אנליטית בהמשך.
כיצד זה עובד?
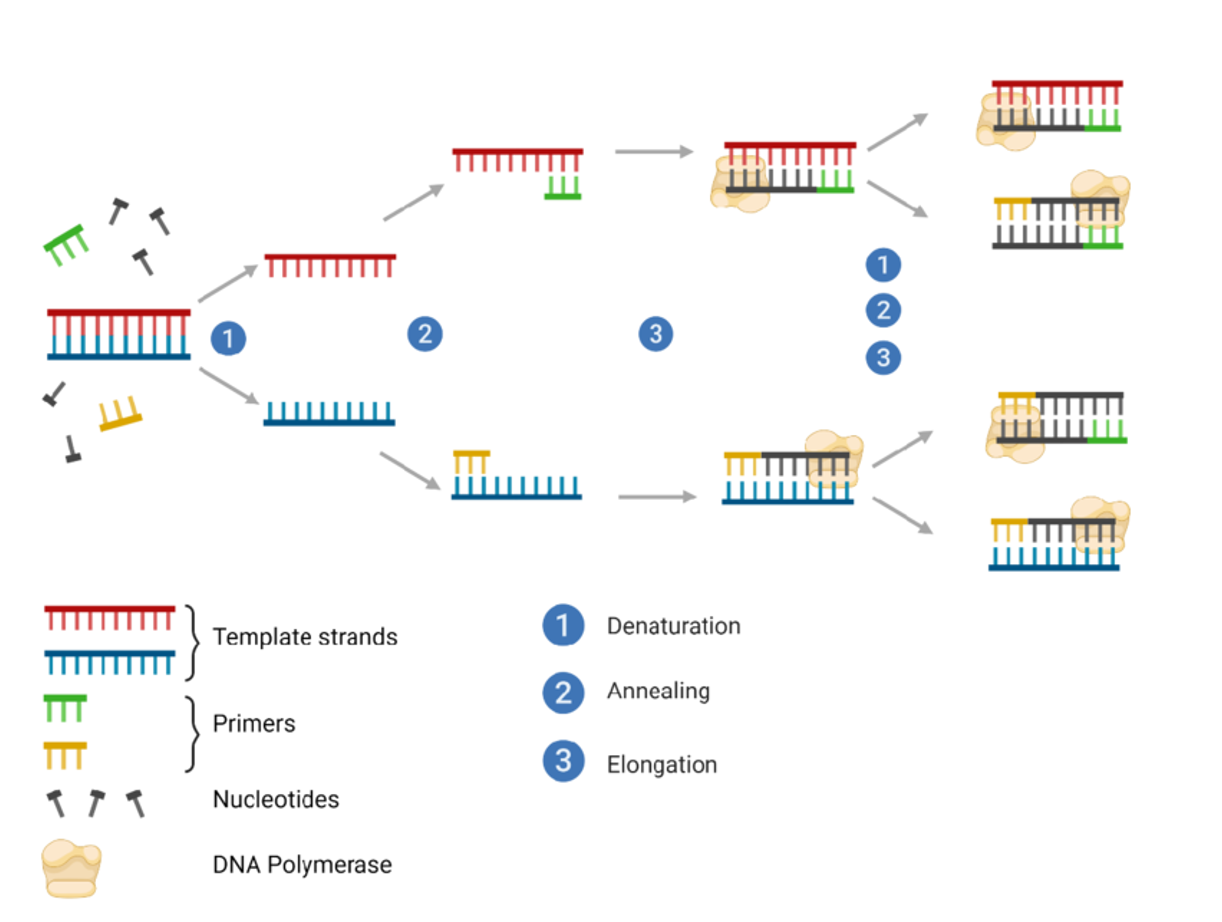
התהליך דומה מאוד לשכפול DNA טבעי, ומורכב משלושה שלבים מחזוריים:
- Denaturation - חימום ה־DNA לצורך הפרדת שני הגדילים
- Annealing - קירור המאפשר היצמדות של primers לרצף המטרה
- Extension - Taq polymerase מאריך את הגדילים החדשים
המחזורים חוזרים מספר רב של פעמים, ובכל מחזור כמות המקטע מוכפלת. לאחר כ־30 מחזורים מתקבלים מיליוני עותקים של המקטע המבוקש.
Primers - רכיב מפתח ב־PCR
שכפול DNA ללא primer אינו אפשרי: ה־polymerase אינו מתחיל סינתזה מאפס, אלא דורש רצף קצר שמשמש בתור נקודת התחלה.
מה שייחוד ב־PCR הוא שה־primers מתוכננים מראש בהתאם לאזור הרצוי להגברה: נקבע המקטע המטרה, ונבחרים primers הנקשרים אליו באופן ספציפי.
דרישות ל־primer איכותי
- דיוק - התאמה מלאה לרצף המטרה.
- ייחודיות - primer קצר מדי (למשל ATG) יופיע במקומות רבים בגנום ולכן עלול להיקשר לאתרים רבים. נדרש primer ארוך מספיק כדי להיות ייחודי סטטיסטית.
- שני כיוונים - נדרשים Forward primer ו־Reverse primer, אחד מכל צד של המקטע. כך מוגבר האזור שביניהם, ובמקרים מסוימים מתאפשר גם ריצוף משני הכיוונים.
בעיה קלאסית: Pseudogenes
בבדיקות מסוימות קיימת מגבלה משמעותית עקב pseudogenes — רצפים בגנום שדומים מאוד לגן האמיתי אך אינם פונקציונליים.
הקושי הוא ש־primer עלול להיקשר ל־pseudogene במקום לגן האמיתי. במצב כזה תתקבל הגברה של רצף שאינו המטרה, דבר שעלול להוביל לתוצאות שגויות.
חלק ג׳: עבודה עם RNA
למה בכלל RNA אם ה־DNA זהה בכל תא?
נכון — ה־DNA זהה בכל תאי הגוף. הסיבה לעבוד עם RNA היא ש־RNA משקף ביטוי גנים. לא כל גן פעיל בכל תא: תא לב יבטא גנים של חלבוני לב, ותא מוח יבטא גנים הקשורים לנוירוטרנסמיטורים. לכן, כשנדרש לבדוק אם גן מסוים מתבטא ברקמה מסוימת, המידע הרלוונטי נמצא ב־RNA ולא ב־DNA.
מתי בודקים RNA?
- בדיקת ביטוי גנים - האם גן מסוים פעיל ברקמה מסוימת.
- הערכת המשמעות של מוטציות ב־splicing - כאשר קיים שינוי באזור של splice site ורוצים לדעת האם הוא אכן משפיע על תהליך ה־splicing, יש לבדוק את ה־RNA ולראות מה התקבל בפועל.
האתגר: RNA אינו יציב
בניגוד ל־DNA שיכול להישמר לאורך זמן, RNA מתפרק במהירות ובעל זמן מחצית חיים קצר. לכן נדרשת הפקה ועבודה מהירה ומבוקרת.
הפתרון: cDNA
כדי לעבוד עם RNA בצורה נוחה, מבצעים המרה שלו ל־DNA באמצעות האנזים Reverse Transcriptase (מוכר מווירוסים כמו HIV).
תהליך העבודה:
mRNA → Reverse Transcription → output: cDNA (complementary DNA)
המאפיין המרכזי של cDNA הוא שהוא לא מכיל אינטרונים, משום שה־mRNA כבר עבר splicing. לכן cDNA מייצג בפועל את “הגן בלי האינטרונים”.
בשלב הזה ניתן להגביר את ה־cDNA באמצעות PCR רגיל ולהמשיך בעבודה מעבדתית.
חלק ד׳: Gel Electrophoresis - הפרדה לפי גודל
העיקרון
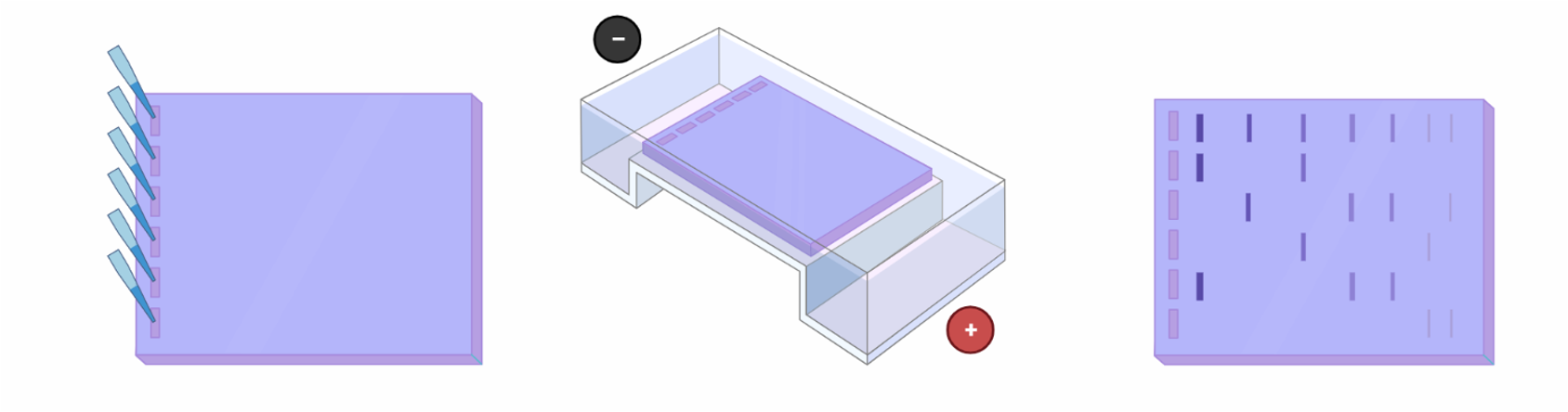
ג׳ל אלקטרופורזה מאפשרת הפרדה של מולקולות לפי גודל ומטען. את הדגימה טוענים על ג׳ל, מפעילים שדה חשמלי, והמולקולות נעות דרכו.
- מולקולות קטנות נעות מהר יותר בג׳ל.
- מולקולות גדולות נעות לאט יותר.
בסיום מתקבלת הפרדה: כל מקטע מופיע במיקום שונה בהתאם לגודלו.
למה זה שימושי?
לאחר PCR ניתן להריץ את התוצר על ג׳ל כדי לבדוק:
- האם בכלל התקבל תוצר
- מה הגודל של התוצר שהתקבל
- האם הגודל שהתקבל תואם את הגודל הצפוי
כמעט תמיד משתמשים גם ב־Ladder (סולם) — תערובת של מקטעים בגדלים ידועים — לצורך השוואה והערכת גדלים.
חלק ה׳: שיטות Blotting - Western, Northern, Southern
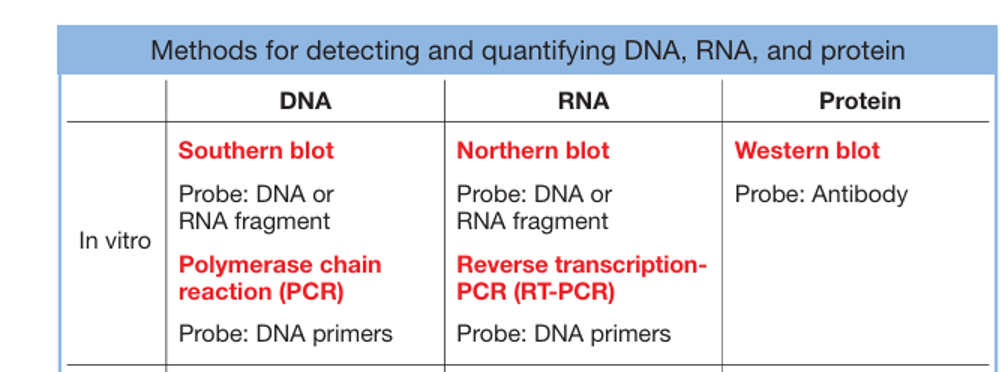
הרעיון הכללי
שיטות ה־Blotting פועלות על עיקרון משותף: הפרדה לפי גודל, העברה לממברנה, ולאחר מכן זיהוי של מולקולה ספציפית באמצעות נוגדן או probe.
שלושת הסוגים
| שיטה | מה נבדק |
|---|---|
| Western Blot | חלבונים |
| Northern Blot | RNA |
| Southern Blot | DNA |
השם Southern נקבע על שם החוקר Edwin Southern. בהמשך, כשפותחו שיטות מקבילות ל־RNA ולחלבונים, נקבעו השמות Northern ו־Western כבדיחה “גיאוגרפית”.
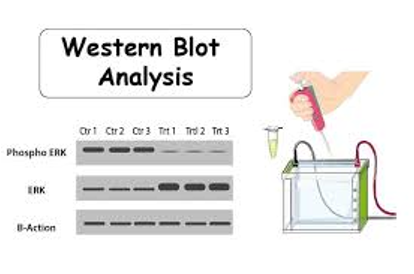
כיצד Western Blot עובד?
- הפקה - הפקת חלבונים מהתאים
- הפרדה - הרצה על ג׳ל; החלבונים נפרדים לפי גודל
- העברה לממברנה - העברת החלבונים מהג׳ל לממברנה ייעודית
- נוגדן ראשוני - הוספת נוגדן ספציפי לחלבון הנבדק
- נוגדן שניוני - הוספת נוגדן שמזהה את הנוגדן הראשוני, והוא פלואורסצנטי
- קריאה - זיהוי פס זוהר במקום שבו נמצא החלבון
למה נדרשים שני נוגדנים?
הנוגדן הראשוני מספק את הספציפיות לחלבון המטרה, אבל הוא לא מסומן ולכן אינו “נראה” ישירות. הנוגדן השניוני נקשר לנוגדן הראשוני (כלומר, “נוגדן נגד נוגדנים”) ומסומן כך שניתן לזהות את מיקום החלבון על הממברנה.
חשיבות ה־Control
הודגש בהרצאה שתמיד נדרש Control.
מקובל להשתמש ב־Housekeeping gene/protein — גן או חלבון שמצויים באופן קבוע בתאים, כמו Actin או GAPDH. מטרת הבקרה היא להבחין בין היעדר ביטוי אמיתי לבין כשל טכני (הפקה לא תקינה, נוגדן שלא עבד, העברה לא טובה וכדומה). כאשר גם ה־housekeeping אינו מופיע, הסבירות הגבוהה היא לתקלה טכנית ולא למסקנה ביולוגית.
בדוגמה שהוצגה בהרצאה נבדק אינסולין בתאי כליה לעומת תאי לבלב: בבקרת housekeeping (למשל RPS7) הופיע פס בשני סוגי התאים, בעוד שבאינסולין הופיע פס רק בלבלב ולא בכליה — תוצאה הגיונית, משום שאינסולין מיוצר בלבלב ולא בכליה.
מה עדיין בשימוש כיום?
- Western Blot - עדיין בשימוש נרחב, בעיקר במחקר, לזיהוי ביטוי של חלבון ספציפי.
- Northern Blot - פחות נפוץ, משום שקיימות שיטות יעילות יותר לבדיקת RNA.
- Southern Blot - כמעט ואינו בשימוש, למעט בדיקה אחת: Fragile X syndrome. במחלה זו קיים אזור של חזרות DNA שמתארך, וריצוף אזורים חזרתיים עלול להיות מאתגר. Southern blot מאפשר להעריך את גודל האזור ובכך מסייע באבחנה.
חלק ו׳: Restriction Enzyme Analysis - זיהוי מוטציות ידועות
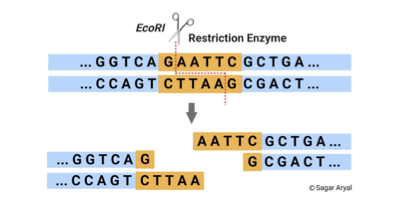
העיקרון - חיתוך DNA ברצפים ספציפיים
אנזימי Restriction (Restriction Enzymes) חותכים DNA ברצפים ספציפיים. לדוגמה, אנזים מסוים יחתוך בכל פעם שיזהה את הרצף GAATTC. כאשר קיימת מוטציה שמשנה את הרצף הזה, ייתכן שהאנזים כבר לא יזהה את האתר — ולכן לא יתרחש חיתוך.
כאשר קיימת מוטציה ידועה (למשל שינוי G ל־C בעמדה מסוימת), ניתן להבדיל בין רצף תקין לבין רצף מוטנטי לפי דפוס החיתוך. לדוגמה:
- רצף תקין: GAATTC (נחתך)
- רצף מוטנטי: CAATTC (לא נחתך)
תהליך העבודה
- הגברת האזור ב־PCR
- הוספת האנזים החותך
- הרצה על ג׳ל
תוצאות אפשריות
- נחתך ← מתקבלים שני פסים קטנים
- לא נחתך ← מתקבל פס אחד גדול
- הטרוזיגוט ← מתקבלים שלושה פסים (אלל אחד נחתך ואלל אחד לא)
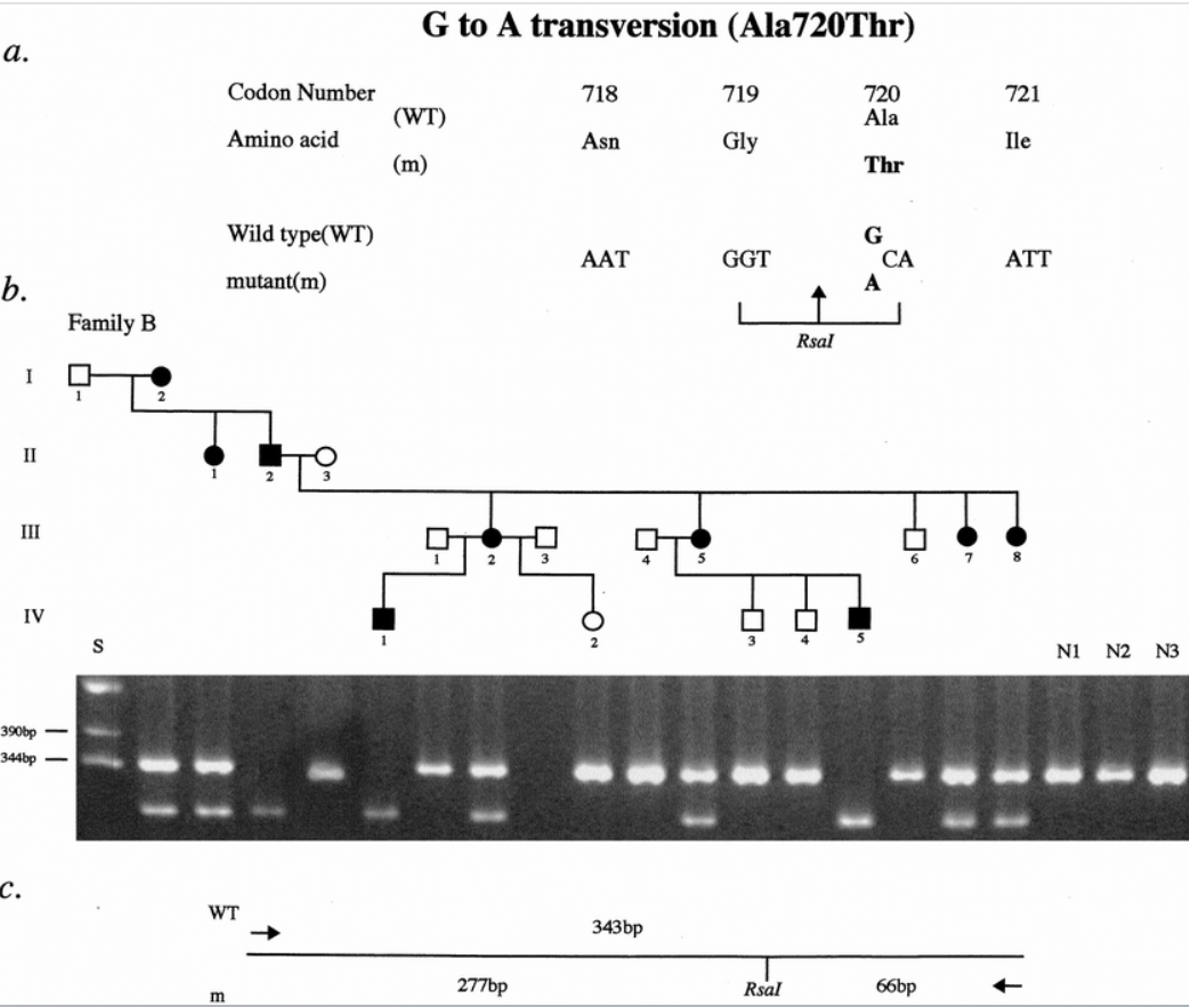
דוגמה קלינית שהוצגה בהרצאה
משפחה עם מחלה רצסיבית:
- ההורים (נשאים) - שלושה פסים: אלל אחד עם מוטציה (לא נחתך) ואלל אחד תקין (נחתך)
- הילד החולה - פס אחד: שני האללים עם מוטציה (לא נחתכים)
מתי משתמשים ב-Restriction Enzyme Analysis?
השיטה מתאימה רק כאשר המוטציה ידועה מראש; לא ניתן לגלות בעזרתה מוטציות חדשות.
שימוש נפוץ הוא Population Screening: כאשר מזוהה מוטציה חדשה באוכלוסייה/יישוב מסוים ונדרש להעריך שכיחות. ריצוף מאות דגימות הוא מאמץ גדול, בעוד ש־PCR + אנזים חיתוך + ג׳ל עבור מספר רב של דגימות הוא תהליך פשוט יותר, זול ומהיר.
נקודה חשובה: תלוי בתכנון
הודגש בהרצאה שייתכן מצב שבו המוטציה נחתכת והתקין לא, או להפך — הכול תלוי ברצף הספציפי ובאנזים שנבחר. בשאלות מבחן המידע הדרוש לכך יינתן במפורש.
דוגמה משיעורי הבית
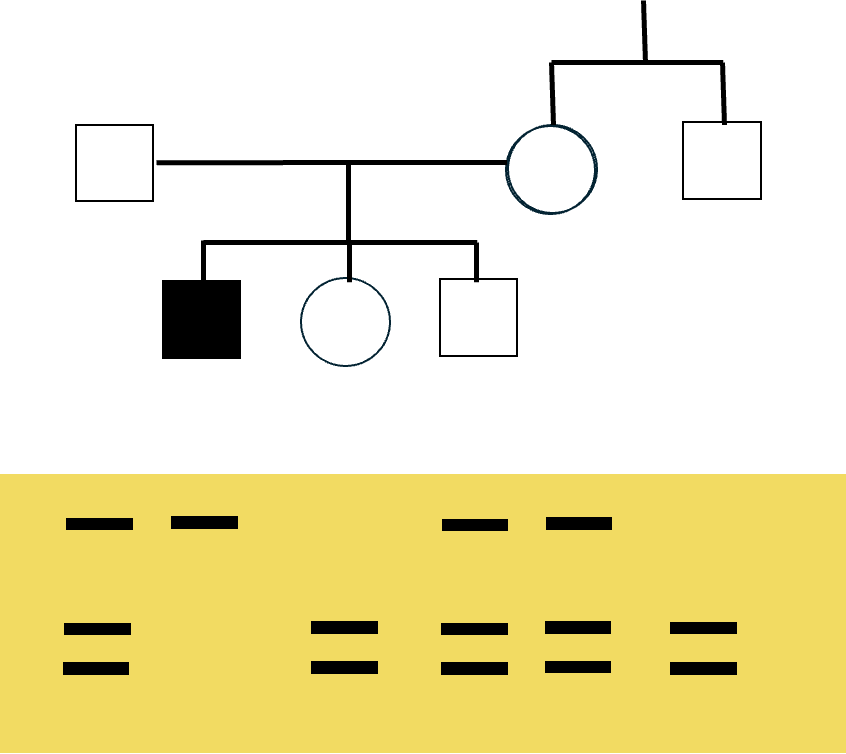
- האבא (שמאל מעלה) - שלושה פסים מכאן שהוא נשא כי אלל אחד נחתך והשני לא. נסמן N אלל תקין ו m מוטנטי, אז הגנוטיפ של האב יהיה N/m. ההסבר הוא פשוט ששני פסים מייצגים אתר חיתוך, אז יש אתר חיתוך ועוד אלל נוסף.
- הבן החולה (שמאל למטה) - אין אתר חיתוך אז פס אחד, כלומר m/m (האלל לא נחתך). חולה רצסיבי.
- הבת - לא נשאית. יש שני פסים אז N/N כי האתר נחתך ומכאן שיש אתר חיתוך.
- הבן הימני (עם שלושת הפסים) - דומה לאבא, הוא נשא עם גנוטיפ של N/m כמפורט לעיל.
- האמא - גם כן נשאית, מאותם שיקולים הגנוטיפ שלה יהיה N/m.
- הדוד (עם שני פסים) - בדומה לבת הגנוטיפ יהיה N/N, הוא לא נשא.
חלק ז׳: Sanger Sequencing - הקלאסיקה
רקע
Sanger Sequencing הייתה שיטת הריצוף המרכזית לגנים לפני עידן ה־NGS. השיטה פותחה בשנות ה־70 על ידי פרדריק סנגר וזיכתה אותו בפרס נובל. גם Human Genome Project השלים את ריצוף הגנום האנושי באמצעות Sanger — פרויקט עצום שנמשך שנים.
העיקרון הגאוני
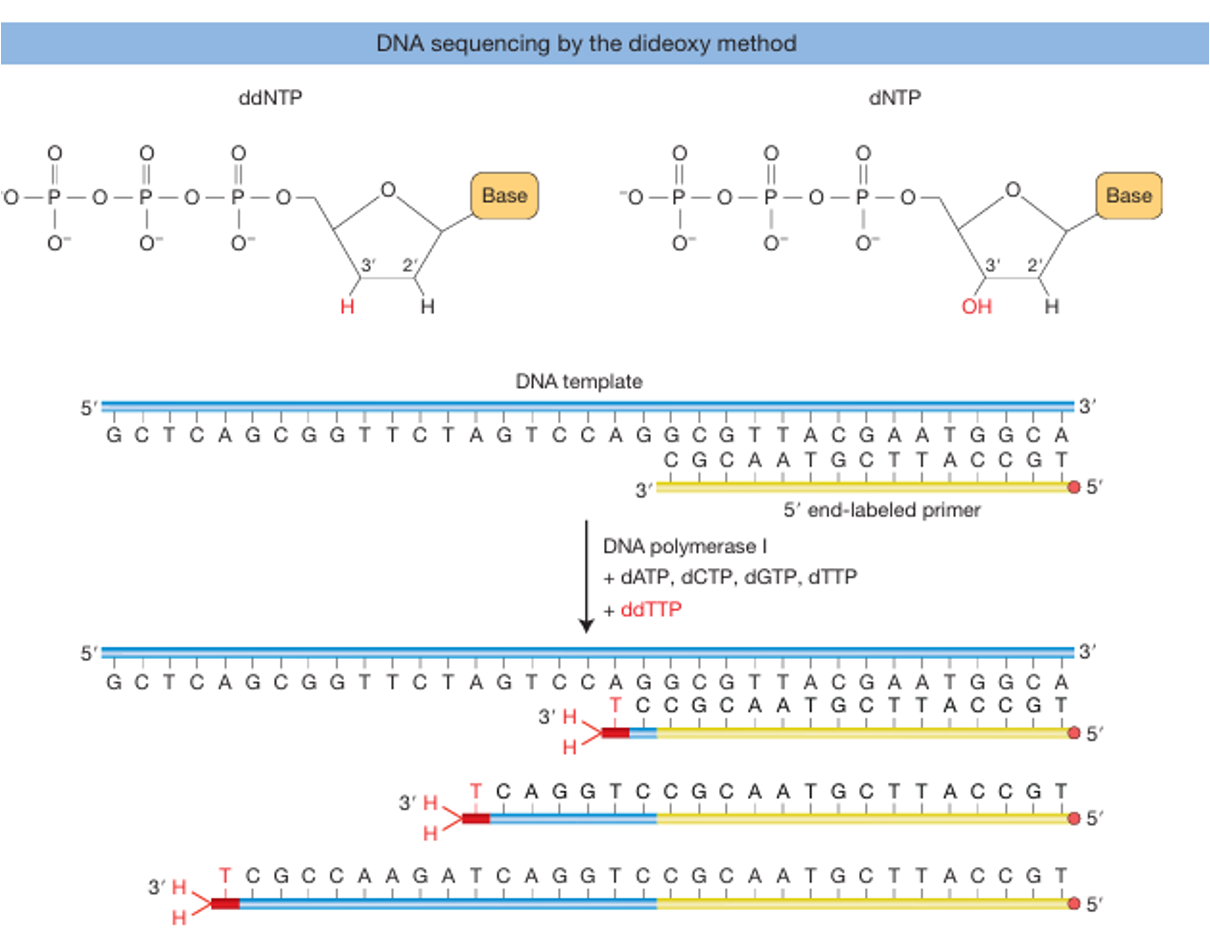
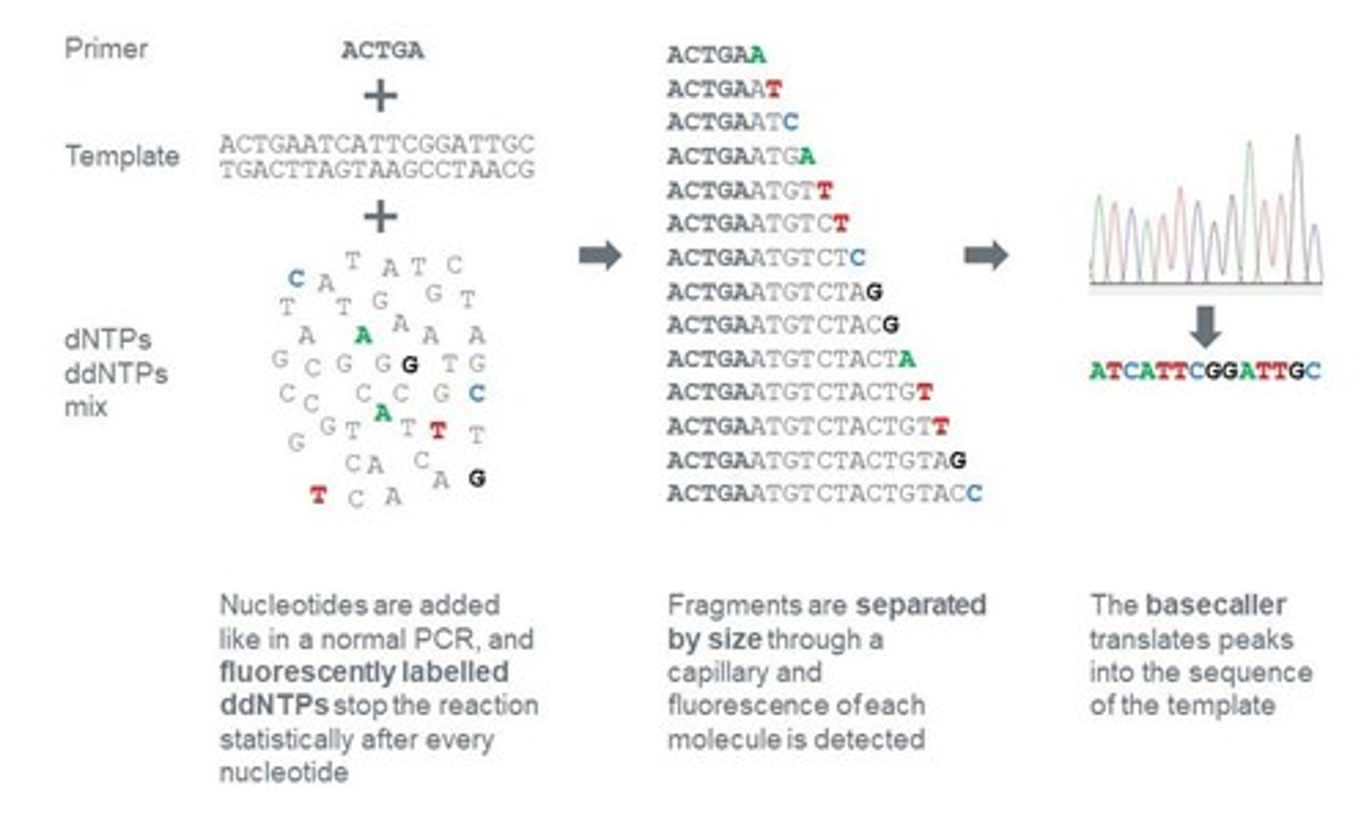
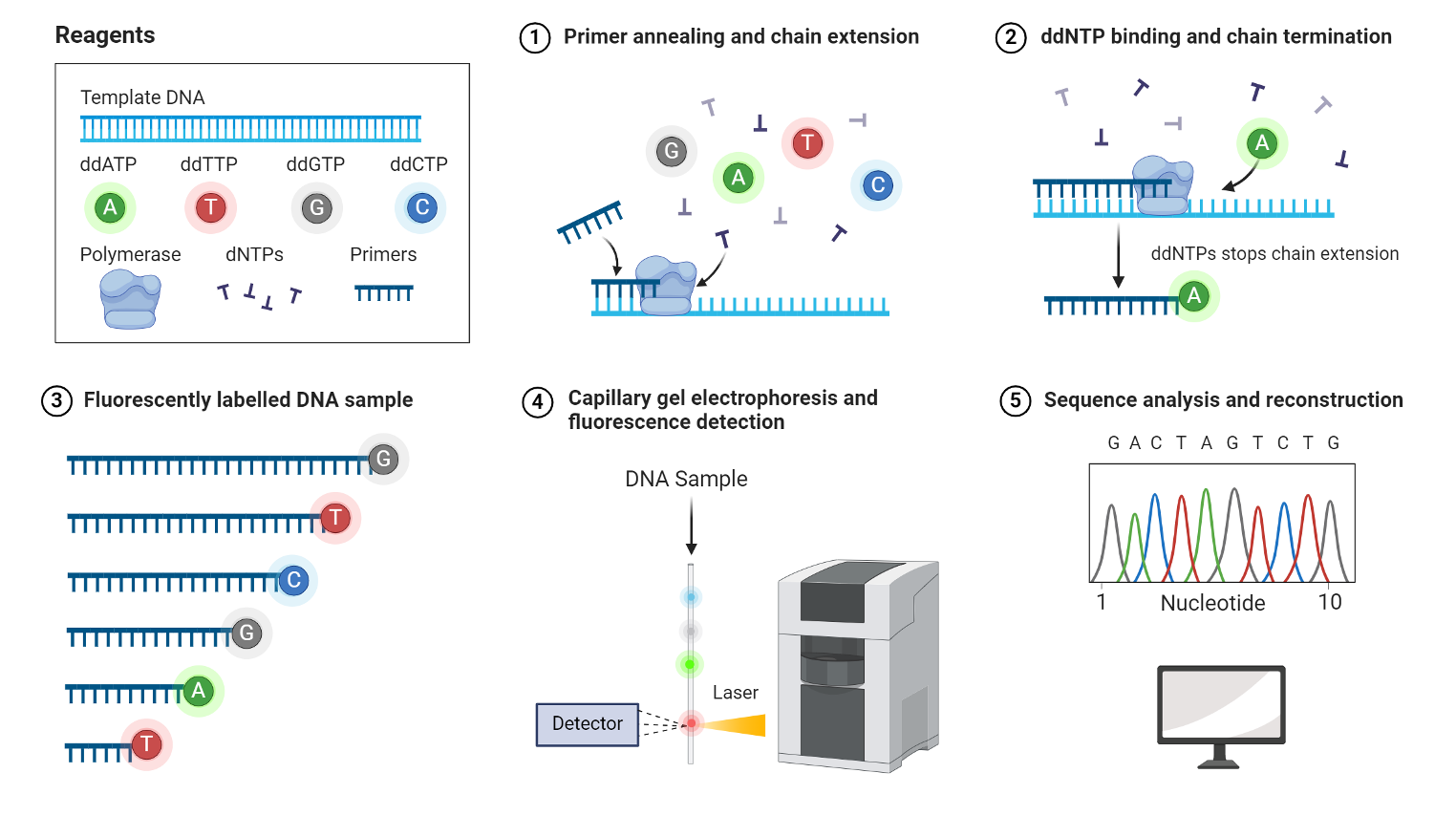
בריאקציית PCR רגילה משתמשים בנוקלאוטידים תקינים (dNTPs) שמאפשרים לשרשרת להמשיך להתארך.
בשיטת Sanger מוסיפים בנוסף גם ddNTPs (dideoxy nucleotides) — נוקלאוטידים “פגומים” שחסרה בהם קבוצת OH. כאשר ddNTP משתלב בשרשרת, ההתארכות נעצרת ולא ניתן להוסיף אחריו נוקלאוטידים נוספים.
מה מתרחש בפועל?
מערבבים dNTPs עם כמות קטנה של ddNTPs. באופן אקראי, לעיתים ddNTP נכנס לשרשרת ועוצר אותה. לאחר ביצוע הריאקציה פעמים רבות מתקבל אוסף של מקטעים בכל האורכים האפשריים, משום שהעצירה מתרחשת כל פעם בעמדה אחרת.
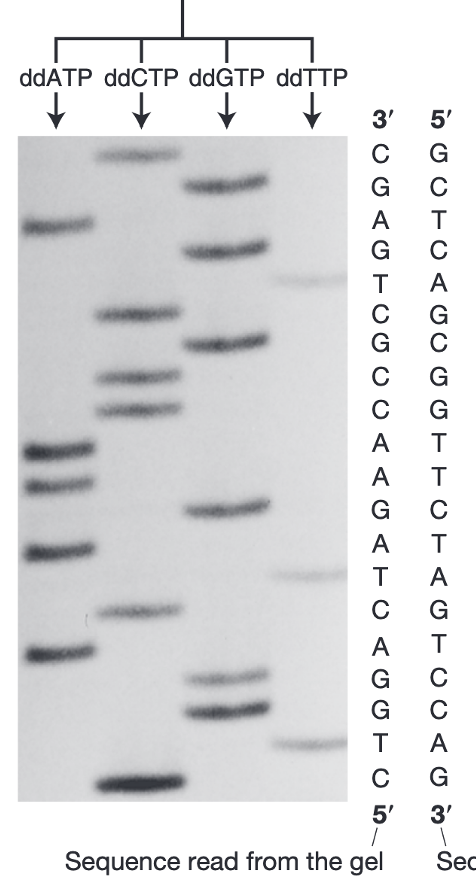
איך מתקבל הרצף?
כל אחד מארבעת ה־ddNTPs (A, T, C, G) מסומן בצבע שונה. את תוצרי הריצוף מריצים על ג׳ל (או בקפילרה), שם הם נפרדים לפי גודל. עבור כל אורך מזוהה הצבע הזוהר, והוא קובע מהו הבסיס באותה עמדה.
 |  |
דגש: בריצוף בשיטת סנגר מתחילים מריאקציית pcr, כלומר מרצפים קטע קטן יחסית של דנ״א (עד 1000 בסיסים) מתוך מקטע דנ״א שהוגבר.
הוזכר בהרצאה שבעבר הקריאה נעשתה ידנית מתוך הג׳ל (“כאן A, כאן T…”), אבל כיום התהליך מתבצע באופן אוטומטי (למעט בשיעורי הבית שקיבלנו).
מגבלות
- אורך מוגבל - ניתן לרצף בערך כ־500 בסיסים בריאקציה אחת; מעבר לכך איכות הקריאה יורדת.
- עבודה ידנית - נדרש תכנון primers לכל אקסון, ביצוע PCR וריצוף. גן עם 72 אקסונים (כמו NF1) משמעותו לפחות 72 ריאקציות נפרדות.
- זמן ועלות - ריצוף גן שלם דורש זמן רב.
מתי עדיין משתמשים ב־Sanger?
- מוטציות ידועות - כאשר ידוע בדיוק מה מחפשים, Sanger ממוקד יכול להיות מהיר וזול יותר מ־NGS.
- אישור ממצאים - לעיתים מאמתים ממצאים שהתקבלו ב־NGS באמצעות Sanger.
- בדיקות משפחתיות - לאחר זיהוי מוטציה במשפחה, ניתן לבדוק קרובים בצורה נוחה באמצעות Sanger.
דוגמה משיעורי הבית: פיענוח תוצאות ריצוף בג׳ל

הרצף שהתקבל בג׳ל:
- 3’- AGATTTCCAGGTCCCCAGTGGAAAGCTT -5’
- 5’ - TCTAAAGGTCCAGGGGTCACCTTTCGAA -3’
חלק ח׳: Next Generation Sequencing - המהפכה
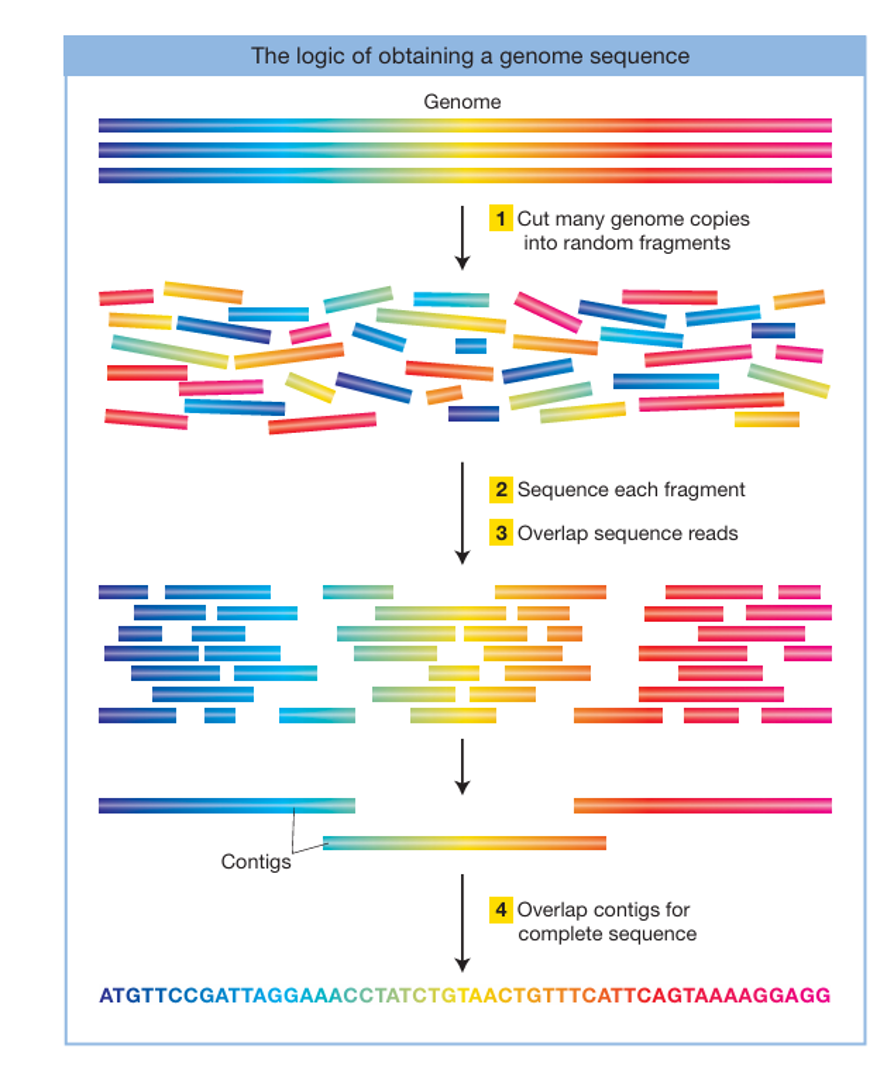
מה נשתנה?
במקום לרצף מקטע אחד בכל פעם, NGS מאפשר ריצוף של מיליוני מקטעים במקביל — שינוי שהפך את התחום כולו.
העיקרון - ארבעה שלבים מרכזיים
- שבירה - חיתוך ה־DNA למקטעים קצרים
- Capture - שימוש ב־probes (כמו הרבה primers) כדי “לתפוס” את האזורים המיועדים לריצוף
- ריצוף מקבילי - המכשיר מרצף את כל המקטעים בו־זמנית
- Alignment - המחשב מסדר את המקטעים לפי הסדר הנכון, בדומה להרכבת פאזל
Alignment - סידור הפאזל
לאחר הריצוף מתקבלים מיליוני מקטעים קצרים. כדי לדעת מאיפה כל מקטע הגיע, משתמשים ב־Reference sequence — רצף ייחוס מוכר של הגנום האנושי (מפרויקט Human Genome וממאגרי מידע גדולים של אנשים בריאים).
האלגוריתם משווה כל מקטע לרצף הייחוס ומאתר את המיקום המתאים לו — כמו התאמת חתיכת פאזל לתמונה השלמה.
חפיפה מסייעת: המקטעים אינם זהים באורכם, ולעיתים קיימת חפיפה בין מקטעים סמוכים. החפיפה מחזקת את הוודאות שהסידור נכון.
Coverage ו־Depth - מדדי איכות
- Coverage - האם כל האזור הרצוי אכן רוצף. ייתכן שאזור מסוים לא “נתפס” על ידי ה־probes או שהוא בעייתי מבחינה כימית. אזור שאינו מכוסה אינו מאפשר הסקת מסקנות לגביו.
- Depth - כמה פעמים כל עמדה נרוצפה. קריאה בודדת יכולה להיות טעות; לעומת זאת, קריאה חוזרת (למשל 30 פעמים) מעלה משמעותית את רמת הביטחון.
הוזכר בהרצאה כי הסטנדרט המינימלי הוא לפחות 20 קריאות כדי לקבוע בביטחון את הבסיס בעמדה מסוימת.
כיצד מזהים הטרוזיגוטיות?
אם בעמדה מסוימת מתקבלות למשל 50% קריאות של T ו־50% קריאות של C, הדבר מתאים להטרוזיגוטיות: אלל אחד עם T ואלל אחד עם C. אם מתקבלות 100% קריאות של T, מדובר בהומוזיגוטיות.
סוגי בדיקות NGS
- Panel - ריצוף רשימת גנים מוגדרת מראש. לדוגמה: “פאנל ראסופתיות” הכולל את הגנים הידועים ל־Noonan ולמחלות דומות.
- Whole Exome Sequencing (WES) - ריצוף כל האקסום (האזור המקודד). זה בערך 1% מכל ה־DNA, אך בו נמצאות רבות מהמוטציות הגורמות למחלות.
- Whole Genome Sequencing (WGS) - ריצוף הגנום כולו: אקסונים, אינטרונים ואזורים בין־גניים.
כמה וריאנטים מתקבלים?
בריצוף אקסום לאדם מתקבלים בערך 100,000 וריאנטים — כ־100,000 עמדות שבהן הרצף שונה מה־reference.
רובם המכריע הם וריאציות נורמליות שמייצגות שונות בין אנשים ואינן גורמות למחלה. האתגר הוא לזהות מתוך עשרות אלפי השינויים את השינוי הבודד שגורם למחלה.
אתגרים
- אזורים חזרתיים - רצפים כמו CAGCAGCAGCAG מקשים על קביעה מדויקת של מספר החזרות, משום שהמקטעים דומים מאוד.
- אזורי GC - אזורים עשירים ב־G וב־C בעייתיים מבחינה כימית ולכן קשים לריצוף.
- Pseudogenes - קושי דומה ל־PCR: מקטע יכול “להתאים” ליותר ממיקום אחד בגנום בשל דמיון גבוה בין רצפים.
חלק ט׳: Variant Classification - הלב של הגנטיקה הקלינית
האתגר המרכזי
זוהה שינוי בגן — השאלה המיידית היא מה המשמעות שלו: האם מדובר בשינוי גורם־מחלה, או בשונות תקינה שלא קשורה למחלה?
זאת אחת השאלות המרכזיות בגנטיקה קלינית. בהרצאה הודגש שרוב הגנטיקה עוסקת ב־Variant Classification.
סולם הסיווג (ACMG Guidelines)
| סיווג | משמעות | מה עושים עם זה |
|---|---|---|
| Pathogenic | גורם מחלה (>99% ודאות) | מדווחים, משפיע על טיפול |
| Likely Pathogenic | כנראה גורם מחלה (~90%) | מדווחים, מתייחסים כמו Pathogenic |
| VUS | לא ידוע | מדווחים, אך לא פועלים על סמך זה |
| Likely Benign | כנראה שפיר (~90%) | לרוב לא מדווחים |
| Benign | שפיר (>99%) | לא מדווחים |
VUS - Variant of Unknown Significance
VUS אינו אבחנה. בהרצאה הודגש כי לעיתים קיימת אי־הבנה: עצם הופעת “שינוי גנטי” בדו״ח אינה אומרת שמדובר באבחנה. משמעות VUS היא “לא ידוע” — אין מסקנה האם השינוי גורם למחלה או לא.
למרות זאת, VUS עשוי להיות מדווח משום שייתכן שבעתיד יתווסף מידע: יימצאו אנשים נוספים עם אותו שינוי, או יתפרסם מחקר שיאפשר סיווג מחדש.
כיצד מסווגים? איסוף ראיות
הסיווג מבוסס על גישה של Evidence-Based Medicine: נאספות ראיות בעד ונגד פתוגניות, לכל ראיה ניתן משקל/ניקוד, ולבסוף מתקבלת הכרעה מסכמת.
ראיות חזקות לסיווג וריאנטים
1. שכיחות באוכלוסייה - הראיה החזקה ביותר
כאשר שינוי מופיע בשכיחות גבוהה באוכלוסייה, הוא כנראה לא יכול להיות הסבר למחלה נדירה וקשה. למשל, שינוי שנמצא אצל 50% מהאנשים לא יכול להיות גורם למחלה מונוגנית נדירה — אחרת מחצית מהאוכלוסייה הייתה חולה בה (והיא לא הייתה כל כך נדירה).
כלל אצבע: אם שינוי מופיע ביותר מ־0.5% מהאוכלוסייה, הוא כנראה לא גורם למחלה מונוגנית קשה.
מאגרי מידע לשכיחות:
- gnomAD - המאגר הגדול והמרכזי, כולל מאות אלפי אקסומים וגנומים
- ExAC - גרסה קודמת
- 1000 Genomes - המאגר המקורי
בפועל, ניתן להזין את השינוי ב־gnomAD ולבדוק כמה נשאים קיימים.
2. Loss of Function בגן שפועל ב־Haploinsufficiency
Loss of Function (LoF) הם שינויים שמנטרלים את פעילות הגן, למשל:
- Nonsense - יצירת stop codon מוקדם
- Frameshift - הוספה/מחיקה שמסיטה את מסגרת הקריאה
- מחיקה גדולה - חסר של חלק גדול מן הגן או של הגן כולו
כשהגן פועל במנגנון של Haploinsufficiency (נדרשים שני עותקים תקינים), וריאנט LoF נחשב ראיה חזקה מאוד לפתוגניות.
משקל בסיווג: 8 נקודות - Very Strong evidence.
3. De Novo - הופעה בילד, היעדרות בהורים
שינוי שמופיע בילד חולה ולא קיים בשני הורים בריאים נחשב לראיה חזקה, משום שהוא התרחש ספונטנית ובמקביל הופיעה מחלה.
משקל בסיווג: 4 נקודות - Strong evidence.
חשוב: גם הכיוון ההפוך משמעותי. אם שינוי מורש מהורה בריא, זו ראיה חזקה לשפירות — במיוחד במחלות דומיננטיות קשות.
4. מוטציה שכבר תוארה
כשאותו שינוי בדיוק (או שינוי אחר באותו קודון שמוביל לאותה החלפת חומצת אמינו) כבר תועד בספרות כפתוגני — מדובר בראיה חזקה.
5. עבודות מעבדתיות (Functional Studies)
מחקרים שבודקים בפועל את השפעת השינוי (למשל Western blot, מחקרי RNA ועוד) וכוללים ממצאים כמו חלבון שאינו מיוצר או שאינו מתפקד בצורה תקינה, מספקים ראיה תומכת.
ראיות בינוניות וחלשות
מיקום בחלבון
האם השינוי נמצא באזור חשוב: binding site, דומיין פונקציונלי וכדומה. שינוי באתר קריטי נחשב חשוד יותר.
שימור אבולוציוני
אם חומצת האמינו במיקום זה שמורה לאורך האבולוציה (מדג ועד אדם), קיימת אינדיקציה לחשיבות פונקציונלית ולכן שינוי במיקום כזה עשוי להיות משמעותי יותר.
תוכנות פרדיקציה
כלים כמו SIFT, PolyPhen, CADD מנסים לחזות אם שינוי מזיק על בסיס מבנה החלבון ושימור אבולוציוני.
עם זאת, הודגש בהרצאה: לא להסתמך רק על תוכנות (Softwares) — הן לא תמיד מדויקות, והן מהוות ראיה אחת מתוך מכלול.
תהליך העבודה בפועל
שלב 1: מתקבלים כ־100,000 וריאנטים
לאחר Exome מתקבלת רשימה גדולה במיוחד של וריאנטים.
שלב 2: סינון ראשוני
בוצע סינון על ידי ביואינפורמטיקאים, למשל לפי:
- שכיחות (הסרת וריאנטים שכיחים)
- פנוטיפ (מיקוד בגנים רלוונטיים)
- סוג השינוי (העדפה ל־LoF)
שלב 3: מעבר לרשימה קצרה
בדרך כלל מתקבלים כ־10 וריאנטים שדורשים התייחסות.
שלב 4: סיווג כל וריאנט
נערכת בחינה משותפת של ביואינפורמטיקאים ורופאים, לרוב סביב שאלות כמו:
- מה השכיחות באוכלוסייה?
- האם מדובר ב־De Novo?
- מה סוג השינוי?
- האם קיימת ספרות תומכת?
- על מה מצביעות תוכנות הפרדיקציה?
שלב 5: סיכום וקביעת סיווג
נקבע ניקוד שמוביל לסיווג:
- מעל 10 נקודות ← Pathogenic
- 6-9 נקודות ← Likely Pathogenic
- ביניים ← VUS
כלים שימושיים
- Franklin - כלי חינמי ל־data mining שמציע סיווג על בסיס חיפוש במאגרים
- Varsome - כלי דומה
- ClinVar - מאגר וריאנטים שסווגו בעבר
עם זאת הודגש בהרצאה: בדיקה עצמאית הכרחית — הכלים לא תמיד מדויקים.
חלק י׳: מקרים קליניים
מקרה 1: ילדה עם Split Hand Malformation
הצגה: ילדה שנולדה עם יד וכף רגל לא תקינות — “יד שסועה” (Split Hand). מדובר במום מולד מסוים.
הבדיקה: Trio Exome (הילדה ושני ההורים).
הממצא: וריאנט בגן TP63 — גן המוכר כקשור ל־Split Hand.
תהליך הסיווג:
- De Novo (לא מורש מההורים) ✓
- ממוקם באזור שבו מוכרות מוטציות פתוגניות רבות ✓
- נדיר באוכלוסייה ✓
- תוכנות פרדיקציה מצביעות על פגיעה ✓
- תועדו משפחות נוספות עם אותו שינוי ואותו פנוטיפ ✓
מסקנה: Pathogenic — זו ההבחנה.
מקרה 2: ילד עם איחור התפתחותי - Exome תקין
הצגה: ילד בן 4 עם איחור התפתחותי; ההורים בריאים.
הבדיקה: Trio Exome.
הממצא: לא נמצא ממצא מסביר.
מה המשמעות? אין פירוש הדבר שאין בעיה גנטית. המשמעות היא שלא נמצא שינוי מתאים במסגרת הבדיקה. ייתכן למשל:
- מוטציה באזור שלא כוסה
- מוטציה בגן חדש שעדיין אינו מוכר
- ייתכן גם שמדובר בגורם שאינו גנטי (אך אין ודאות)
מקרה 3: ילד עם VUS
הצגה: ילד בן 4 עם איחור התפתחותי ואוטיזם.
הבדיקה: Exome.
הממצא: וריאנט בגן SHANK3 — גן שקשור לאוטיזם ולאיחור התפתחותי.
הבעיה: לאחר סיווג מתקבל VUS — אין די ראיות לקביעה פתוגניות.
מה עושים? הממצא מדווח, תוך הסבר ברור למשפחה שמדובר בממצא שאינו אבחנה, וייתכן שבעתיד יצטבר מידע שיאפשר סיווג מחדש.
נקודה שהודגשה בהרצאה: קיימת נטייה לפרש “שינוי בגן שקשור לפנוטיפ” כאבחנה, אך זו Selection Bias — וריאנטים שעולים לדיון הם מלכתחילה כאלה שנמצאים בגנים שנבחרו בגלל הפנוטיפ. עצם ההתאמה לקטגוריית הגנים לא מוכיחה שהווריאנט הוא גורם המחלה.
חלק י״א: Secondary Findings ו־Incidental Findings
Secondary Findings - ממצאים משניים
מה זה?
מדובר ברשימה של 84 גנים שנבדקים באופן אקטיבי בכל בדיקת Exome או Genome, גם כאשר אינם קשורים לשאלה הקלינית המקורית.
למה עושים את זה?
משום שמדובר בגנים Actionable — כלומר, אם מתגלה בהם מוטציה קיימת משמעות מעשית ויש מה לעשות מבחינת מניעה/מעקב/טיפול.
דוגמאות:
- BRCA1/2 - סרטן שד ושחלות: מעקב הדוק ואפשרות לניתוח מניעתי.
- גנים של Lynch syndrome - סרטן מעי: קולונוסקופיות בתדירות גבוהה.
- גנים לאריתמיות - סיכון למוות לבבי פתאומי: אפשרויות טיפול מניעתי.
כללים חשובים
- הסכמה מראש: נשאלים מראש האם קיימת רצון לקבל מידע על Secondary Findings.
- דיווח בילדים: לא מדווחים בילדים ממצאים של מחלות “מבוגרים בלבד”. לדוגמה, אם מתגלה BRCA בילדה בת 5 — לא מדווחים. הנימוק: זכות לאוטונומיה; ההחלטה אם לקבל מידע כזה אמורה להתקבל בגיל 18, ולא על ידי ההורים.
חריג: כאשר הממצא רלוונטי לגיל הילדות (למשל APC שיכול לגרום לפוליפים כבר סביב גיל 12) — כן מדווחים.
דוגמה: APC ושתי מוטציות שונות
- מוטציה קלאסית ב־APC - גורמת ל־Familial Adenomatous Polyposis. לפי הדוגמה, 100% מהנשאים יפתחו סרטן מעי, ומתחילים מעקב מגיל 8. זה מוגדר Actionable ולכן מדווחים.
- מוטציה I1307K ב־APC - מעלה סיכון פי 2 בלבד (מ־2% ל־4%). לפי הדוגמה, אין שינוי מעשי במעקב (ממילא קיימת המלצה לכלל האוכלוסייה לקולונוסקופיה מגיל 50), ולכן זה מוגדר לא Actionable ולא מדווחים.
Incidental Findings - ממצאים מקריים
אלו ממצאים שעולים במקרה, ללא חיפוש יזום.
דוגמה: נבדק גן הקשור לפרכוסים, ובמהלך הניתוח מזוהה במקרה מוטציה בגן הקשור לחירשות — ממצא שאינו קשור לשאלה המקורית אך התגלה.
גם כאן, לפי ההרצאה, הנוהל הוא לשאול מראש האם קיימת רצון לקבל מידע על ממצאים מסוג זה.
חלק י״ב: סוגיות אתיות
מקרה לדוגמה: BRCA ב־Panel ממוקד
המקרה: ילדה קטנה מקהילה חרדית עם חשד ל־Noonan syndrome. הוזמן Panel ממוקד שכולל רק גנים של Noonan, במטרה להימנע מחשיפה למידע נוסף שעלול להפריע לשידוכים.
מה קרה בפועל: המעבדה עדכנה שבמהלך העבודה “בטעות” נצפה שלילדה יש BRCA, אך הוסיפה: “זה לא יופיע בדו״ח”.
הדילמה:
- המידע לא התבקש על ידי הילדה או הוריה
- חשיפתו תפגע באוטונומיה שלהם
- מנגד, מדובר במידע שעשוי להציל חיים בעתיד
הפעולה שנעשתה: האם הוזמנה והומלץ לה לבצע בדיקת BRCA משום שהיא אשכנזייה (כהמלצה כללית לנשים אשכנזיות). לאחר חתימה על טופס, היא סירבה להיבדק.
התוצאה: הצוות הרפואי יודע שלילדה יש BRCA אבל הוא לא יכול לעשות דבר על בסיס המידע. ייתכן שבעתיד הילדה תבחר להיבדק, אך כרגע אין אפשרות ליידע אותה או את משפחתה.
העיקרון: אוטונומיה
גם לילדים קיימות זכויות, ובפרט הזכות להחליט על עתידם הגנטי כאשר יגיעו לגיל שבו ניתן להבין את המשמעות.
כמו שלא ניתן לקחת ילד מהרחוב ולבדוק אותו ל־BRCA, כך גם לא אמור להתאפשר “להשיג” מידע גנטי על ילד דרך בדיקה אחרת באופן עקיף.
חלק י״ג: Rapid Whole Genome - BabySeq
מדובר בריצוף גנום מהיר לתינוקות בטיפול נמרץ, כאשר התשובה מתקבלת בתוך 5 ימים במקום שבועות.
למי זה מיועד?
לתינוק שנולד בזמן ונראה תקין, אך לאחר מכן חלה הידרדרות פתאומית — פרכוסים, כשל נשימתי, בעיות מטבוליות — ללא הסבר ברור.
למה Genome ולא Exome?
- מהיר יותר - מדלג על שלב ה־Capture
- כיסוי טוב יותר של האקסום
- מאפשר לזהות גם ממצאים מחוץ לאקסום
מקרה חיובי: הצלחה
- הצגה: תינוק בן 48 שעות עם פרכוסים רציפים.
- ממצא: מוטציה בגן הקשור ל־B6-Dependent Epilepsy — מצב שבו הפרכוסים נפסקים לאחר מתן ויטמין B6.
- תוצאה: ניתן ויטמין B6, והתינוק חזר להיות תקין — חיים ניצלו.
מקרה שלילי: גם זה עוזר
הצגה: תינוק עם פרכוסים קשים בדומה למקרה הקודם. ממצא: מוטציה בגן הקשור ל־Epileptic Encephalopathy — מחלה עם פרכוסים עמידים לטיפול, ללא אפשרות טיפולית יעילה. תוצאה: הופסק ניסיון בטיפולים אגרסיביים לאחר שהובהר שלא צפויה תועלת. הדבר סייע להורים להתמודד ולקבל תחושת סגירת מעגל.
המסר
גם כאשר אין טיפול, לאבחנה יש ערך. היא עשויה לסייע ב־
- הפסקת בירור מיותר וכואב
- מתן תשובות להורים
- תכנון הריונות עתידיים
- קבלת החלטות לגבי המשך או הפסקת טיפול
חלק י״ד: מושגים חשובים נוספים
Locus Heterogeneity
מצב שבו מוטציות בגנים שונים גורמות לאותה מחלה.
דוגמה קלאסית: Noonan syndrome Noonan יכול להיגרם ממוטציות בגנים שונים, למשל:
- PTPN11
- SOS1
- RAF1
- KRAS
- ועוד גנים נוספים
ההסבר הוא שכל הגנים הללו משתתפים באותו מסלול ביולוגי — RAS-MAPK pathway. פגיעה בנקודות שונות באותו מסלול יכולה להוביל לתמונה קלינית דומה.
RASopathies
קבוצת מחלות הנגרמות ממוטציות במסלול RAS-MAPK, ובהן:
- Noonan syndrome
- Costello syndrome
- CFC (Cardio-Facio-Cutaneous)
- Neurofibromatosis type 1
למחלות אלה יש מאפיינים חופפים, כגון קומה נמוכה, מומי לב, ו־דיסמורפיזם פנים.
Noonan syndrome - “ה־Turner האוטוזומלי”
מאפיינים אופייניים:
- קומה נמוכה
- צוואר רחב
- אוזניים נמוכות ומסובבות
- מום לבבי
ההבדל לעומת Turner:
- Turner - 45,X (חסר כרומוזום X)
- Noonan - תורשה אוטוזומלית (מוטציה בגן)
- Turner - מום באאורטה, למשל Coarctation
- Noonan - מום במוצא הריאה, למשל Pulmonic stenosis
סיכום נקודות למבחן
מה חשוב לזכור
- שכיחות באוכלוסייה היא הראיה החזקה ביותר לסיווג וריאנטים: וריאנט שכיח אינו מתאים כמסביר מחלה נדירה.
- De Novo במחלה דומיננטית נחשב ראיה חזקה מאוד לפתוגניות.
- Loss of Function בגן Haploinsufficient הוא ממצא חזק מאוד (8 נקודות).
- VUS אינו אבחנה — המשמעות היא “לא ידוע”.
- Coverage = האם האזור רוצף; Depth = כמה פעמים כל עמדה נקראה.
- Exome ≈ 1% מהגנום (האזור המקודד בלבד); Genome כולל את כל הרצף.
- Secondary Findings = רשימת 84 גנים Actionable שנבדקים באופן יזום.
- BRCA בילדים לא מדווח מטעמי אוטונומיה, כדי לאפשר החלטה עצמאית בגיל 18.
- הבדלים בין שיטות:
- Western blot = חלבונים;
- Northern = RNA;
- Southern = DNA.
- Sanger מוגבל לכ־~500 bp, בעוד NGS מרצף מיליוני מקטעים במקביל.
מה יכול להופיע בשאלה
- וריאנט עם נתונים (שכיחות, האם De Novo, סוג שינוי) ושאלה האם הסיווג מתאים ל־Pathogenic
- השוואה בין סוגי בדיקות (Panel / Exome / Genome, או Sanger מול NGS)
- שאלות על דיווח: מתי מדווחים ומתי לא
- מושגים מרכזיים: Coverage, Depth, VUS
נאמר בהרצאה ש“זה לא יהיה מסובך”: המידע הדרוש צפוי להינתן בשאלה, והדגש הוא על הבנת העקרונות. מניסיון, עדיף לשנן כמה שיותר, במיוחד פרטים קטנים וטכניים.
דור פסקל