חזקים ב-RNA וחלבונים?
נסו 10 שאלות בנושא, עם עדיפות לשאלות שלא הצלחתם. בסיום תקבלו ציון וסקירה על הטעויות שלכם. הצלחתם? גלו בחנים בדפים נוספים
תוצאות המבחן
היסטוריית מבחנים
טוען את השאלות...
אנא המתן...
הבהרה מבואית: כיוון סינתזת חומצות הגרעין
לפני התעמקות בתהליכי עיבוד ה-RNA, הכרחי להבהיר עיקרון יסודי בביולוגיה מולקולרית. כל הפולימרזים המעורבים בסינתזת חומצות גרעין - RNA פולימרז I, II ו-III, וכן DNA פולימרז - מסנתזים תמיד בכיוון מ-5’ ל-3’. הסיבה לכך נעוצה במנגנון הפעולה של הפולימרזים: הם יכולים להוסיף נוקלאוטידים חדשים אך ורק לקצה 3’-OH של הנוקלאוטיד הקודם.
בהקשר זה, שני הגדילים של ה-DNA מקבלים שמות שמבוססים על הכיוון שלהם יחסית לכיוון השעתוק:
- גדיל Sense: גדיל ה-DNA המכוון מ-5’ ל-3’
- גדיל Antisense: הגדיל המשלים (קומפלמנטרי), המכוון מ-3’ ל-5’
מאחר שה-RNA פולימרז יכול לסנתז רק בכיוון 5’ ל-3’, השעתוק מתחיל תמיד מהגדיל Antisense, המשמש כתבנית (template) לסינתזת ה-RNA.
עיבוד mRNA: שלושה תהליכים מרכזיים
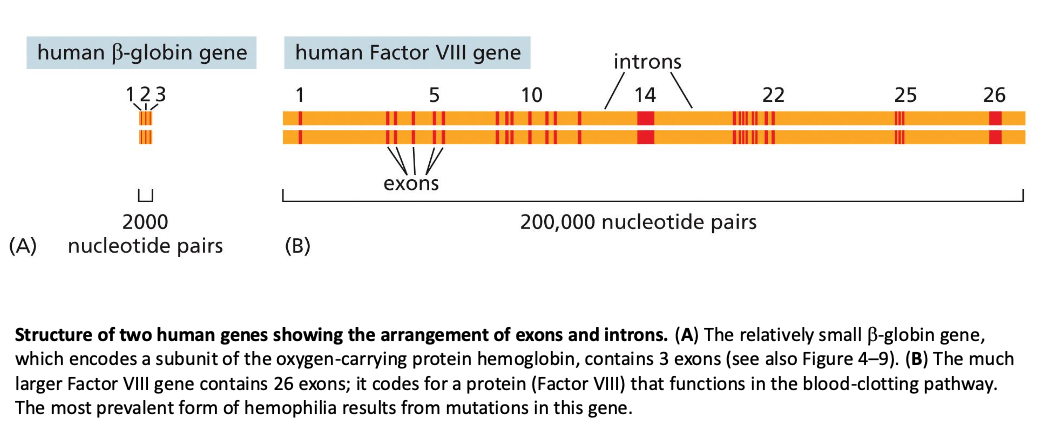
רקע כללי על עיבוד RNA
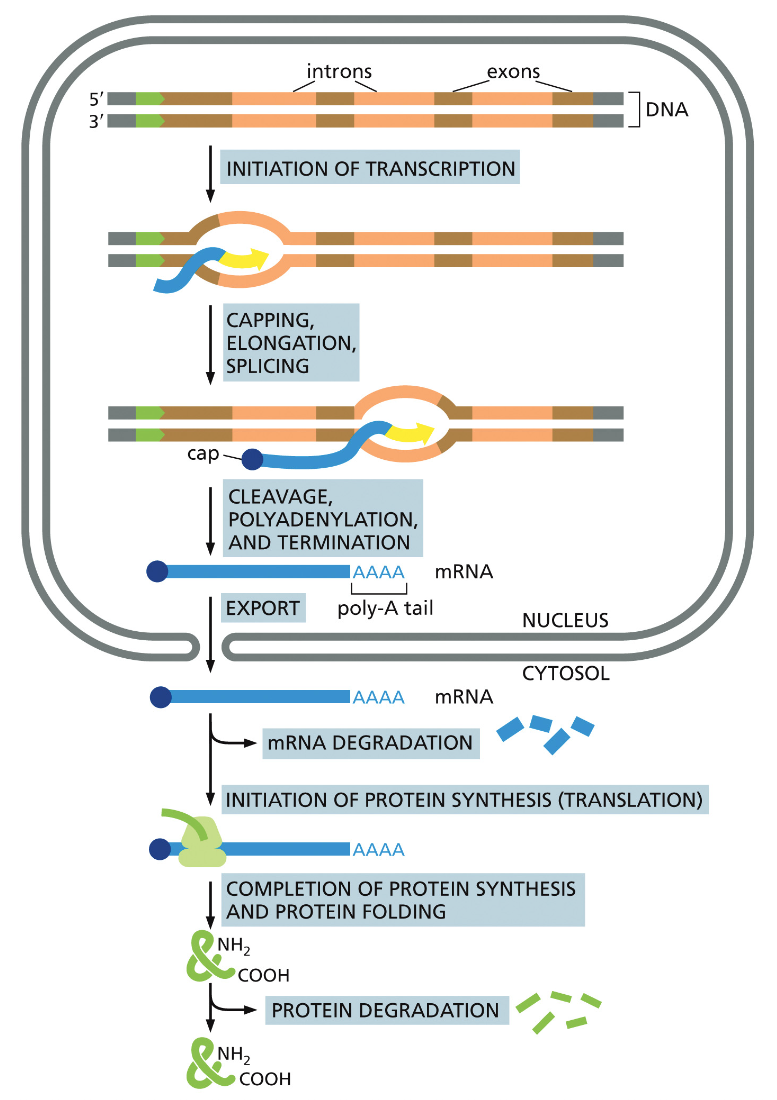
במהלך השעתוק בתאים איקריוטיים, ה-RNA החדש שנוצר אינו מוכן מיד לתרגום. התוצר
הראשוני, הנקרא pre-mRNA או precursor mRNA, שיוצא על האנטיסנס גדיל ה-DNA,
חייב לעבור שלושה תהליכי עיבוד (processing) מרכזיים המתרחשים כבר במהלך השעתוק עצמו, ולא רק לאחריו. תהליכים אלו הכרחיים להפיכת ה-pre-mRNA ל-mRNA בוגר (mature mRNA) ותפקודי.
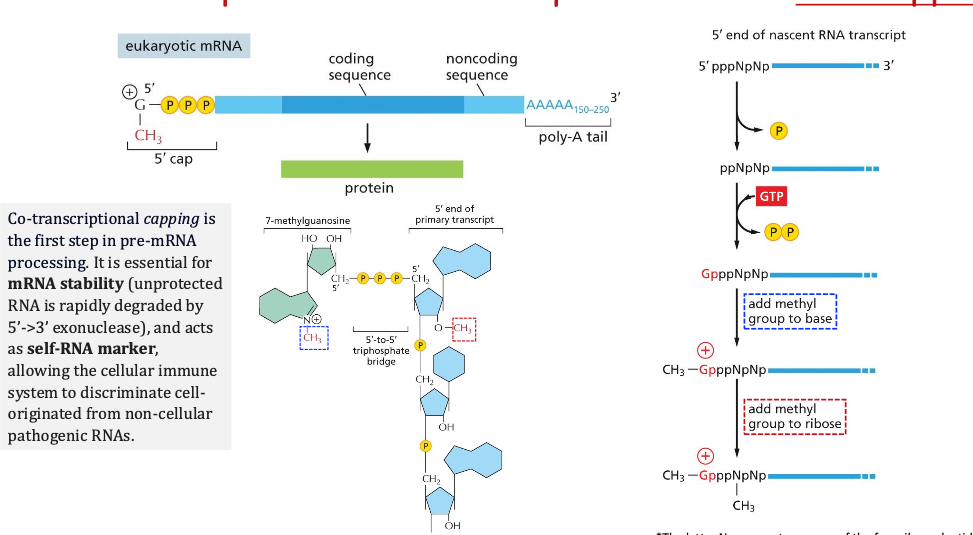
התהליך הראשון: Capping - הוספת הכובע
התהליך הראשון הוא הוספת 7-methylguanosine cap לקצה 5’ של ה-RNA הנוצר. מודיפיקציה זו כוללת הוספת גואנין במהופך (במבנה 5’-5’ במקום 5’-3’ הרגיל) עם קבוצת מתיל על הנוקלאוטיד השני.
$\text{m}^7\text{G}$ -
7-methylguanosine cap
תפקידי ה-Capping:
- הגנה מפני פירוק: הכובע מגן על ה-mRNA מפני אקסונוקלאזות 5’ שמפרקות RNA לא מוגן
- זיהוי על ידי הריבוזום: הכובע משמש אתר זיהוי ספציפי לתת-היחידה הקטנה (40S) של הריבוזום
- הובלה מהגרעין: הכובע נדרש לטרנספורט יעיל דרך הנקבוביות הגרעיניות (nuclear pores)
- התחלת תרגום: הכובע מסמן את נקודת ההתחלה לסריקת הריבוזום
הכובע מתחיל להיווצר כאשר ה-RNA שנוצר עדיין קצר יחסית, מיד עם יציאתו מה-RNA פולימרז.
התהליך השני: Alternative Splicing - הסרת אינטרונים מה-mRNA
לתשומת ליבכם: החלק הבא כולל לא מעט חזרות מיותרות.
השלב השני כולל הסרת רצפים לא מקודדים (אינטרונים) וחיבור הרצפים המקודדים (אקסונים). התהליך מתבצע על ידי מכונת ה-spliceosome - קומפלקס ריבונוקלאופרוטאין מורכב המורכב מ-small nuclear RNAs (snRNAs) וחלבונים נלווים.
החלק המקודד נקרא אקסון (exon), והחלק הלא מקודד נקרא אינטרון (intron).
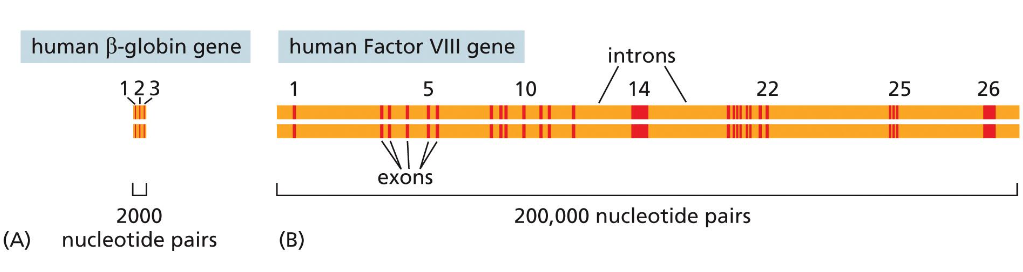
בתמונה, הקווים האדומים הם האקסונים. החיבור שלהם נדרש ליצירת RNA בשל.
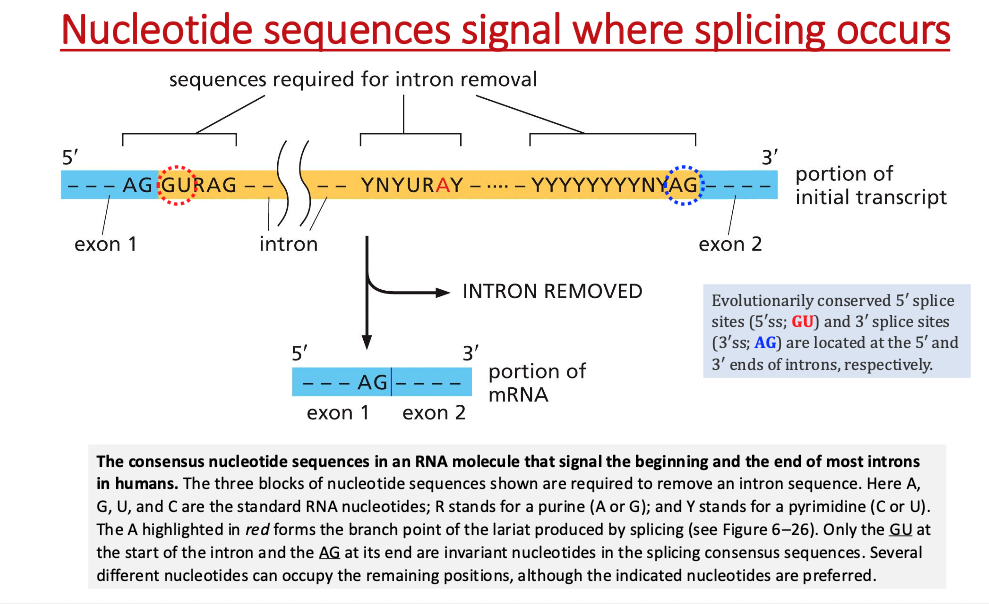
זיהוי גבולות אקסון-אינטרון
השאלה שעולה היא איך מזהים איפה אקסון ואיפה אינטרון.
המידע שנדרש לכך מקודד בתוך ה-DNA - הזיהוי מתבצע על בסיס רצפים קונסרבטיביים (שמורים אבולוציונית):
- כל אקסון מסתיים ברצף A-G
- כל אינטרון מתחיל ברצף G-U ומסתיים ב-A-G
- קיימים רצפים נוספים בתוך האינטרון (כמו branch point sequence) המסמנים למכונת הספלייסינג את נקודות החיתוך המדויקות
ראינו בשלב הזה סרטון שתיאר את פעולת ה-spliceosome ואת תהליך ה-splicing עצמו.
לא קריטי בשלב הזה לזכור את שמות החלבונים המרכיבים את ה-spliceosome, אלא להבין את העיקרון הכללי.
פגיעה בהוצאת האינטרונים משאירה חלבון מוטנטי שיכול לפגוע בתאים. __ סרטן.
השימור האבולוציונית הגבוהה של רצפי האינטרונים מעידה על חשיבותם הקריטית לתפקוד תקין של התא ולהישרדות האורגניזם. מתברר שבתוך האינטרונים יש רצפים שעוברים שיעתוק ויש להם חשיבות רגולטורית, כמו micro RNAs (miRNAs) (ראו בהמשך).
micro RNAs יודעים לזהות mRNAs פגומים, לקשור אותם ולמנוע מהם להתפרק. הם גם יכולים להכווין את ה-mRNA לפירוק על ידי אקסונוקלאזות. או לעצור תרגום.
בנוסף, הם מגוונים את הגנום האנושי ומאפשרים יצירת חלבונים שונים מאותו גן. כלומר חלבונים שונים עם רצפים דומים - איזופורם (isoform).
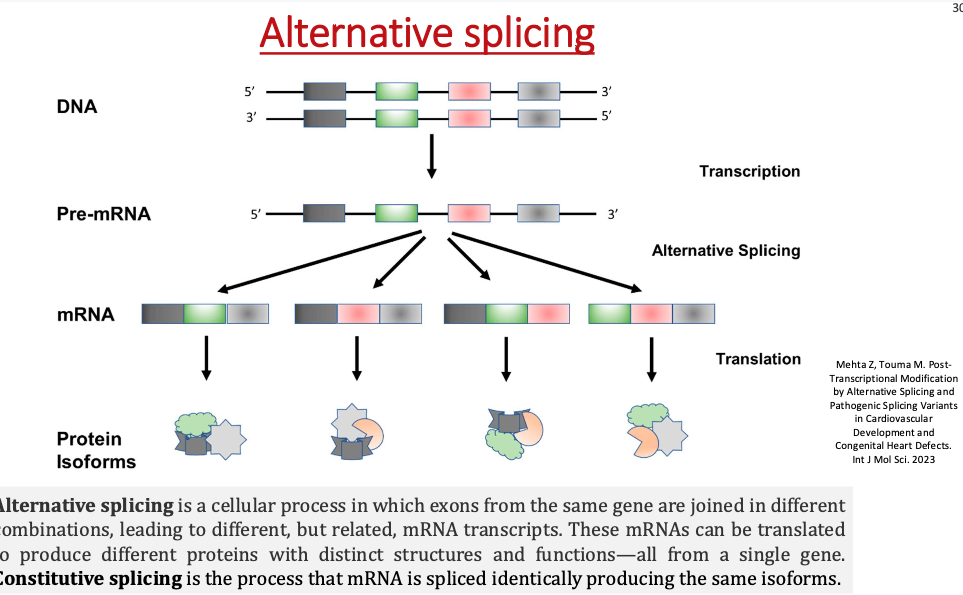
סוגי Splicing:
- Constitutive splicing: הסרה סטנדרטית של כל האינטרונים בצורה קבועה
- Alternative splicing: חיבורים חלופיים בין אקסונים שונים, מה שמאפשר יצירת תוצרים שונים מאותו גן
Alternative Splicing - מנגנון הגיוון החלבוני
המשמעות הביולוגית והאבולוציונית
Alternative splicing מהווה מנגנון מרכזי להסבר הפרדוקס הגנומי: כיצד האדם, עם כ-21,000 גנים מקודדי חלבון בלבד - מספר דומה לזה של זבוב הפירות Drosophila melanogaster או התולעת Caenorhabditis elegans - מצליח לייצר פרוטיאום מורכב הרבה יותר של כ-300,000 חלבונים שונים או יותר.
נתונים כמותיים:
- כ-65% מהגנים האנושיים מכילים אקסונים מרובים ועוברים splicing
- מכ-21,000 גנים העוברים alternative splicing נוצרים כ-300,000 חלבונים שונים
- היחס אינו 1:1 (DNA→RNA→חלבון) אלא 1:רבים (DNA→RNAs מרובים→חלבונים מרובים)
הבסיס המנגנוני
בעוד constitutive splicing מסיר את כל האינטרונים ומחבר את כל האקסונים ברצף (1-2-3-4-5…), alternative splicing מאפשר:
- דילוג על אקסונים (exon skipping): חיבור אקסון 1 ישירות לאקסון 4, תוך דילוג על אקסונים 2-3
- שימוש באתרי splicing חלופיים: שימוש בנקודות חיתוך שונות באותו אקסון
- שמירת אינטרונים: במקרים מסוימים, אינטרון נשמר כחלק מה-mRNA הבוגר
- מודיפיקציות ב-UTRs: שינויים ב-5’ או 3’ UTR המשפיעים על זיהוי וויסות
איזופורמים וספציפיות רקמתית
הגדרת איזופורמים
איזופורמים הם חלבונים הנוצרים מאותו גן אך עם רצפים מעט שונים עקב alternative splicing. החלבונים הללו יכולים לשמר תפקודים בסיסיים משותפים אך לקבל תכונות ייחודיות לרקמה או לתא ספציפיים.
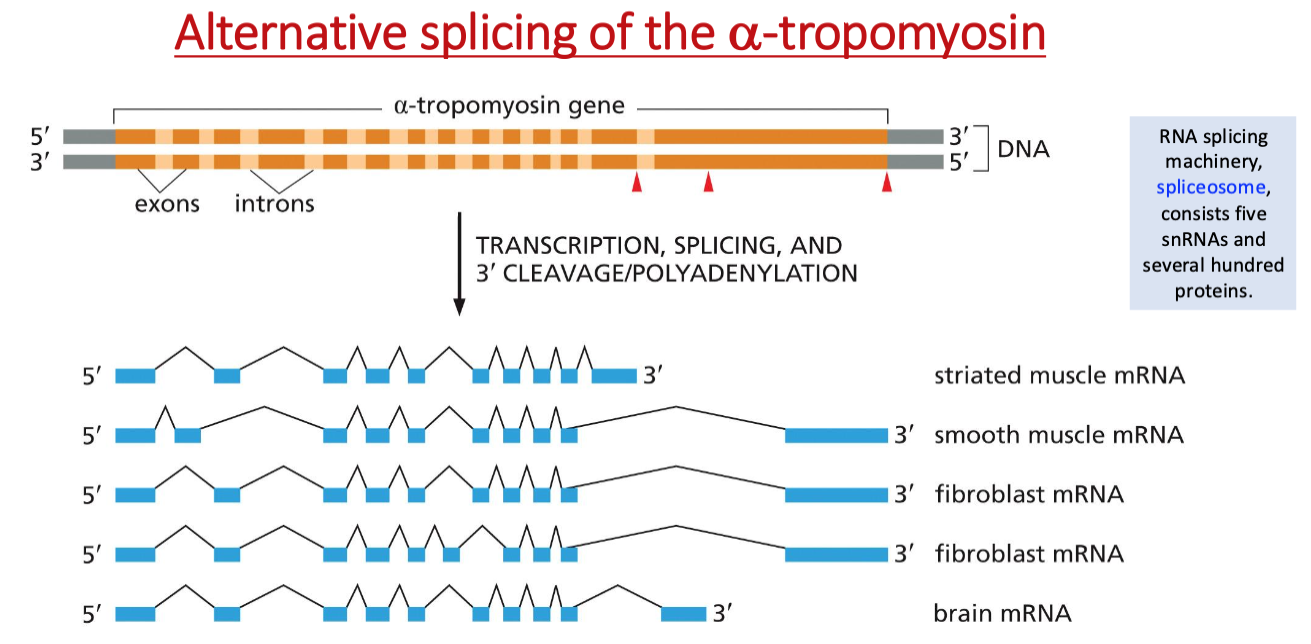
דוגמה: טרופומיוזין (tropomyosin)
טרופומיוזין הוא חלבון המייצב את סיבי האקטין ומבצע alternative splicing ספציפי לרקמה:
- בשריר מפוספף (שלד): איזופורם המתאים לתנועה מהירה ולכיווץ רצוני
- בשריר חלק: איזופורם המותאם לכיווץ איטי ומתמשך
- בפיברובלסטים: איזופורם המיועד לתחזוקת מבנה התא
- במוח ובנוירונים: איזופורם המסייע לארגון הציטוסקלטון בדנדריטים ואקסונים, חיוני לתפקוד הסינפסות
דוגמאות קליניות מרכזיות
פרוגריה (Hutchinson-Gilford Progeria Syndrome)
פרוגריה היא מחלה נדירה שנגרמת על ידי מוטציה דה-נובו (חדשה, לא מתורשת) המשפיעה על הספלייסינג של הגן LMNA. הגן מקודד לחלבונים המארגנים את מעטפת הגרעין ואת מבנה הכרומטין. המוטציה גורמת לספלייסינג לא תקין:
- במקום חיבור תקין של אקסונים 1-2-3-4-5, נוצר חיבור חריג כמו 1-3-5-11
- התוצר הוא חלבון פגום המשבש את מבנה הגרעין
- התוצאה: זדקנות מוקדמת דרמטית - ילדים בני 10-9 נראים כבני 80
דיסטרופין - הגן הגדול ביותר
הגן הגדול ביותר בגנום האנושי עובר alternative splicing נרחב:
- ברקמת השריר בלבד: 12 איזופורמים שונים
- כל איזופורם בעל תפקוד מעט שונה מהאחר
- מוטציות גורמות למחלות שריר קשות כמו Duchenne (דושן) ו-Becker muscular dystrophy
- המחלות פוגעות בעיקר בבנים (הגן נמצא על כרומוזום X)
הוויסות של Alternative Splicing
גורמים שמשפיעים על הספלייסינג:
- מבנה הכרומטין: ארגון שונה של הכרומטין ברקמות שונות משפיע על נגישות למכונת הספלייסינג
- רמות ביטוי של splicing factors: רקמות שונות מבטאות רמות שונות של חלבונים המכווינים את הספלייסינג
- מודיפיקציות אפיגנטיות: מתילציות ואצטילציות המשפיעות על הביטוי והספלייסינג
התהליך עדיין נחקר אך ברור שמדובר בשילוב של גורמים גנטיים, אפיגנטיים וסביבתיים הפועלים יחד.
מיקרו-RNAs וויסות גנים פוסט-טרנסקריפציוני
תפקיד האינטרונים החבוי
אחד הגילויים המרכזיים בעשורים האחרונים היה שאינטרונים אינם “זבל גנטי” כפי שנחשבו בעבר. במקום זאת, הם מכילים רצפים קודדים חשובים למיקרו-RNAs (miRNAs) ו-small nuclear RNAs (snRNAs).
תהליך יצירת miRNAs
- שעתוק: האינטרונים עוברים שעתוק יחד עם שאר הגן
- עיבוד: לאחר הספלייסינג, רצפים ספציפיים באינטרון מעובדים ל-miRNAs
- פירוק חלקי: בעוד רוב האינטרון מתפרק, חלקים ספציפיים נשמרים כmiRNAs פעילים
תפקידי miRNAs
- זיהוי mRNAs פגומים: קישור לרצפים משלימים ב-mRNAs שעברו נזק או שגיאות
- עיכוב תרגום: מניעת גישת הריבוזום ל-mRNA פגום
- הפניה לפירוק: הכוונת mRNAs לא תקינים למנגנוני הפירוק התאיים (degradation)
מנגנון זה מהווה רובד בטיחות חשוב המונע ייצור חלבונים פגומים שעלולים לפגוע בתא או אפילו להוביל למותו.
המשמעות האבולוציונית
השימור האבולוציוני של מערכת ה-miRNAs מעיד על חשיבותה. מערכת זו מאפשרת:
- בקרת איכות: ווידוא שרק mRNAs תקינים מתורגמים
- ויסות דינמי: התאמת רמות החלבונים לצרכי התא
- תגובה למתח: זיהוי מהיר של mRNAs שניזוקו במצבי לחץ
התהליך השלישי: Polyadenylation - הוספת זנב הפולי-A
השלב השלישי כולל הוספת רצף של כ-250 נוקלאוטידי אדנין (פוליאדינין) לקצה 3’ של ה-mRNA. תהליך זה נקרא polyadenylation ומתבצע על ידי אנזימי polyadenylation ספציפיים.
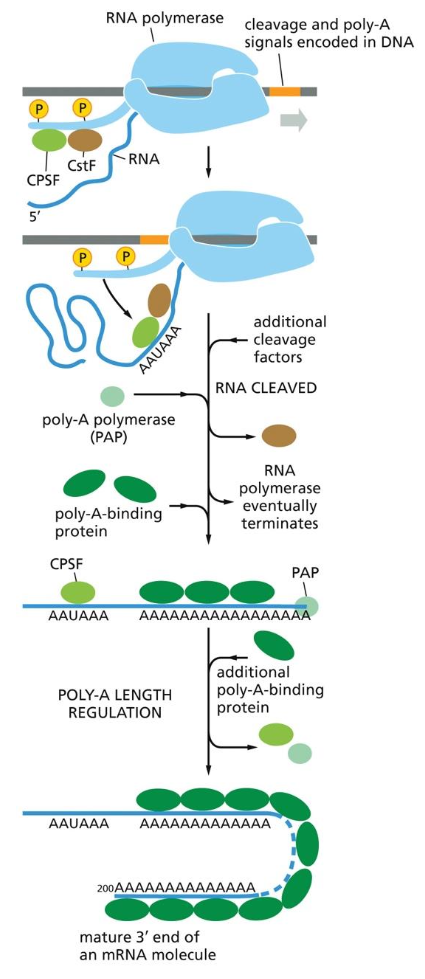
תפקידי ה-Polyadenylation:
- סימן לסיום השעתוק: מסמן ל-RNA פולימרז להשתחרר מה-DNA ולסיים את השעתוק
- יציבות ה-mRNA: הזנב מגן מפני פירוק על ידי 3’ אקסונוקלאזות
- קבלת מבנה תלת-ממדי: מאפשר יצירת מבנה מקופל יציב המסייע לטרנספורט יעיל
- זיהוי על ידי הריבוזום: הזנב נדרש לזיהוי יעיל על ידי חלבוני עזר וליצירת קומפלקס התרגום
- ווסות זמן חיים: אורך הזנב משפיע על יציבות ה-mRNA וזמן החיים שלו
ציור מסכם של שלבי העיבוד ה-mRNA:
ריבוזומל RNA וביוגנזת הריבוזום (rRNAs)
ארגון הגנים והגרעינון
גנים המקודדים ל-rRNA מפוזרים בחמישה כרומוזומים שונים (13, 14, 15, 21, ו-22) אך מתארגנים יחד פיזית בתוך הגרעינון (nucleolus). ארגון מרחבי זה אינו מקרי - הוא מאפשר ייצור יעיל ומהיר של רכיבי הריבוזום על ידי ריכוז כל המשאבים הנדרשים במקום אחד.
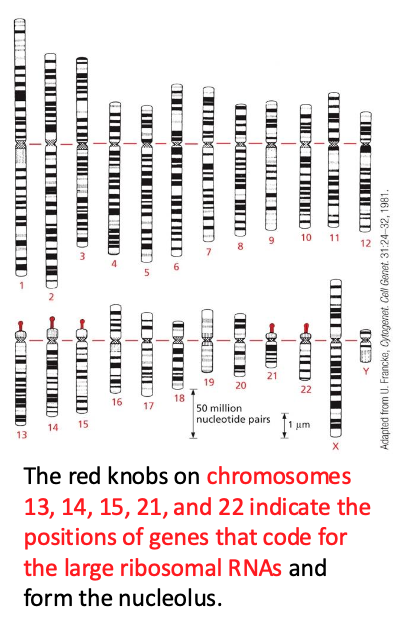
מבנה הגרעינון (nucleolus) - בית חרושת לריבוזומים
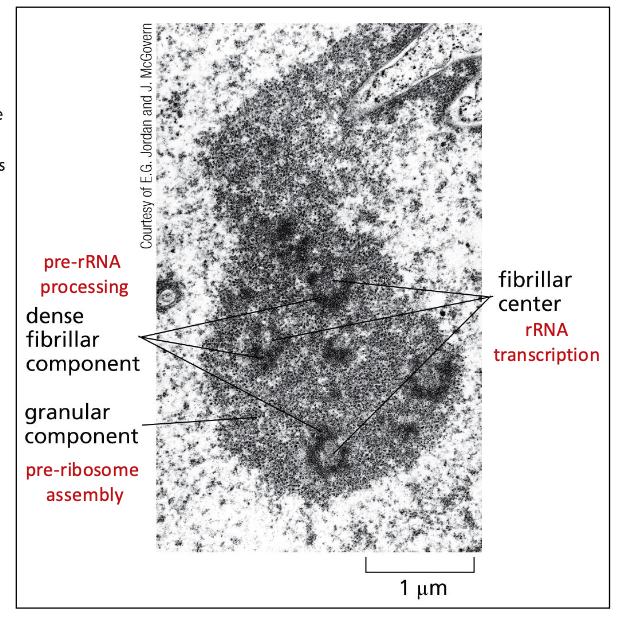
הגרעינון מורכב משלושה תת-מבנים מרכזיים:
- Fibrillar Center: אתר השעתוק הפעיל של גנים שמקודדים ל-rRNA
- Dense Fibrillar Component: אזור העיבוד והמודיפיקציות של ה-rRNA
- Granular Component: אתר ההרכבה הראשונית של תת-יחידות הריבוזום
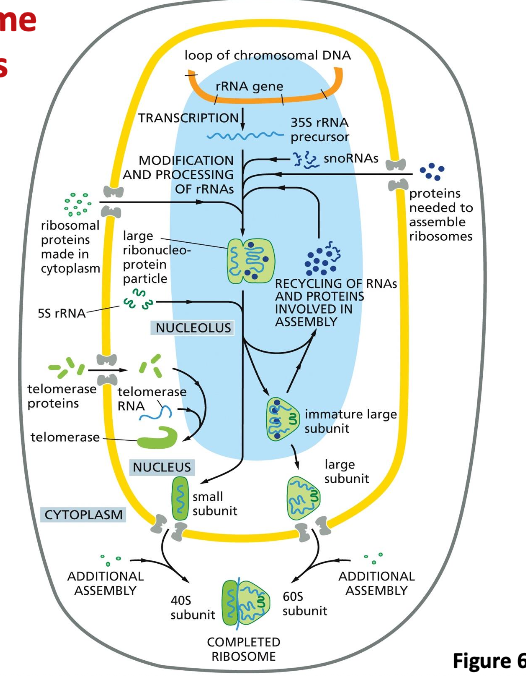
תהליך הביוגנזה המפורט
שעתוק ראשוני
שלושה מארבעת סוגי ה-rRNA (18S, 5.8S, 28S) מסונתזים כמולקולת precursor אחת גדולה (45S pre-rRNA, כ-13,000 נוקלאוטידים) על ידי RNA פולימרז I. ה-rRNA הרביעי (5S) מסונתז בנפרד על ידי RNA פולימרז III.
עיבוד ומודיפיקציות
התהליך כולל מודיפיקציות כימיות מורכבות:
- מתילציות: הוספת קבוצות מתיל לנוקלאוטידים ספציפיים (בעיקר על הריבוז)
- איזומריזציות: המרת אורידין לפסאודואורידין במיקומים מדויקים
- חיתוך מדויק: הפרדה לתת-יחידות בגדלים שונים על ידי אנדונוקלאזות ספציפיות
תפקיד ה-snoRNAs
small nucleolar RNAs מכווינים את תהליכי העיבוד:
- זיהוי אתרים: קישור לרצפים משלימים ב-pre-rRNA
- גיוס אנזימים: הבאת האנזימים המתאימים למיקום המדויק
- הבטחת דייקנות: ווידוא שהמודיפיקציות מתרחשות במיקומים הנכונים בלבד
הרכבת הריבוזום
התהליך מתרחש בשלבים מדורגים:
- עיבוד בגרעינון: מודיפיקציות וחיתוך ראשוני ב-fibrillar ו-dense fibrillar components
- הרכבה חלקית: חיבור עם חלבונים ריבוזומליים יבואים מהציטופלזמה
- יצירת “90S particle”: קומפלקס ביניים גדול הכולל את כל הרכיבים
- large subunit (60S) ו-small subunit (40S) עדיין לא מופרדים
- פיצול לתת-יחידות: חלוקה לתת-יחידה גדולה (~66S) וקטנה (~45S)
- small subunit (40S) מורכבת מ-18S rRNA ו-30 חלבונים
- יצוא לציטופלזמה: מעבר נפרד דרך הנקבוביות הגרעיניות (הגודל הגדול מונע יצוא יחד)
- הרכבה סופית: השלמת המבנה התלת-ממדי והבשלה לתת-יחידות 60S ו-40S
למה תת-יחידות נפרדות?
הנקבוביות הגרעיניות (תעלות ה- nuclear pore) קטנות מדי לאפשר מעבר של ריבוזום שלם. לכן התא פיתח מנגנון של פירוק והרכבה מחדש.
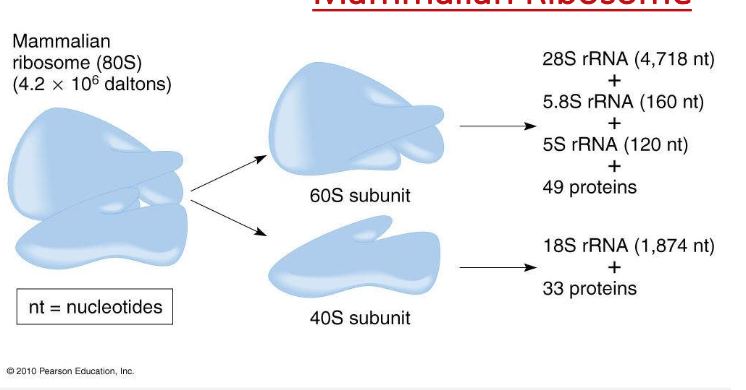
מבנה הריבוזום הבוגר (השלם) ופרס נובל
הריבוזום האיקריוטי השלם (Mammalian Ribosome) (80S) מורכב מ:
- תת-יחידה גדולה (60S): 28S, 5.8S, ו-5S rRNA עם כ-50 חלבונים
- תת-יחידה קטנה (40S): 18S rRNA עם כ-30 חלבונים
הריבוזום הוא שילוב (קומפלקס) של חלבונים ו-rRNA
בשנת 2009, אדה יונת ממכון ויצמן זכתה פרס נובל בכימיה (יחד עם תומאס שטייץ ורמאקרישנן ונקטרמן) על פענוח המבנה התלת-ממדי של הריבוזום וחשיפת מנגנון פעולתו. מחקרם היה פריצת דרך בהבנת אחד המנגנונים המורכבים ביותר בתא.
טרנספר RNA ומנגנון התרגום
מבנה ה-tRNA והתקפלות
מולקולות ה-tRNA (כ-80 נוקלאוטידים) עוברות תהליך התקפלות מורכב ליצירת מבנה תלת-ממדי ייחודי:
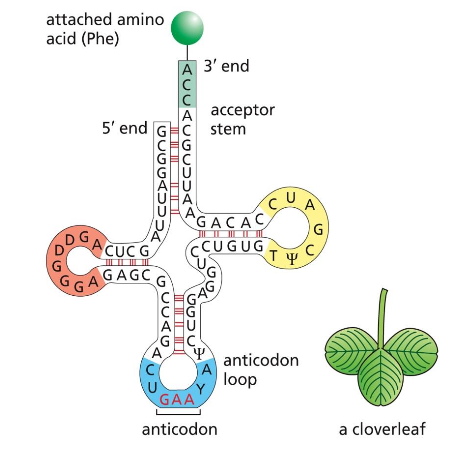
מבנה דו-ממדי (דמוי תלתן)
- קצה 3’ עם רצף CCA שמור: אתר חיבור חומצת האמינו (קונסרבטיבי בכל ה-tRNAs)
- לולאת האנטי-קודון: שלושה נוקלאוטידים המזהים את הקודון ב-mRNA
- לולאות נוספות: D loop, TψC loop המייצבות את המבנה
מבנה תלת-ממדי (דמוי L)
המבנה התלת-ממדי האמיתי דמוי האות L, כאשר:
- חומצת האמינו נמצאת בקצה של זרוע אחת
- האנטי-קודון נמצא בקצה של הזרוע השנייה
- המבנה מייצב על ידי קשרי מימן בין-מולקולריים
מודיפיקציות כימיות
ה-tRNA עובר מודיפיקציות כימיות נרחבות לאחר השעתוק:
- מתילציות: הוספת קבוצות מתיל לבסיסים ולריבוז
- איזומריזציות: המרת אורידין לפסאודואורידין
- מודיפיקציות נוספות: יצירת בסיסים נדירים כמו inosine
מודיפיקציות אלו חיוניות ליציבות המבנה ולתפקוד הנכון.
אין כל כך Caping ו-Polyadenylation ב-tRNA, אלא רק ב-mRNA.
טעינת ה-tRNA על ידי Aminoacyl-tRNA Synthetases
תהליך הטעינה מבוצע על ידי משפחת אנזימים ייחודית (אמינואצילים-tRNA סינתטאזות) שמבצעת זיהוי כפול מדויק:
שלב הזיהוי הראשון - חומצת האמינו
כל aminoacyl-tRNA synthetase מזהה חומצת אמינו ספציפית אחת בלבד על בסיס:
- גודל ומבנה השרשרת הצדדית
- מטען חשמלי
- יכולת יצירת קשרי מימן
שלב הזיהוי השני - tRNA
זיהוי ה-tRNA המתאים מבוסס על:
- רצף האנטי-קודון הספציפי
- מבנה תלת-ממדי ייחודי
- רצפים נוספים ב-tRNA המכונים “identity elements”
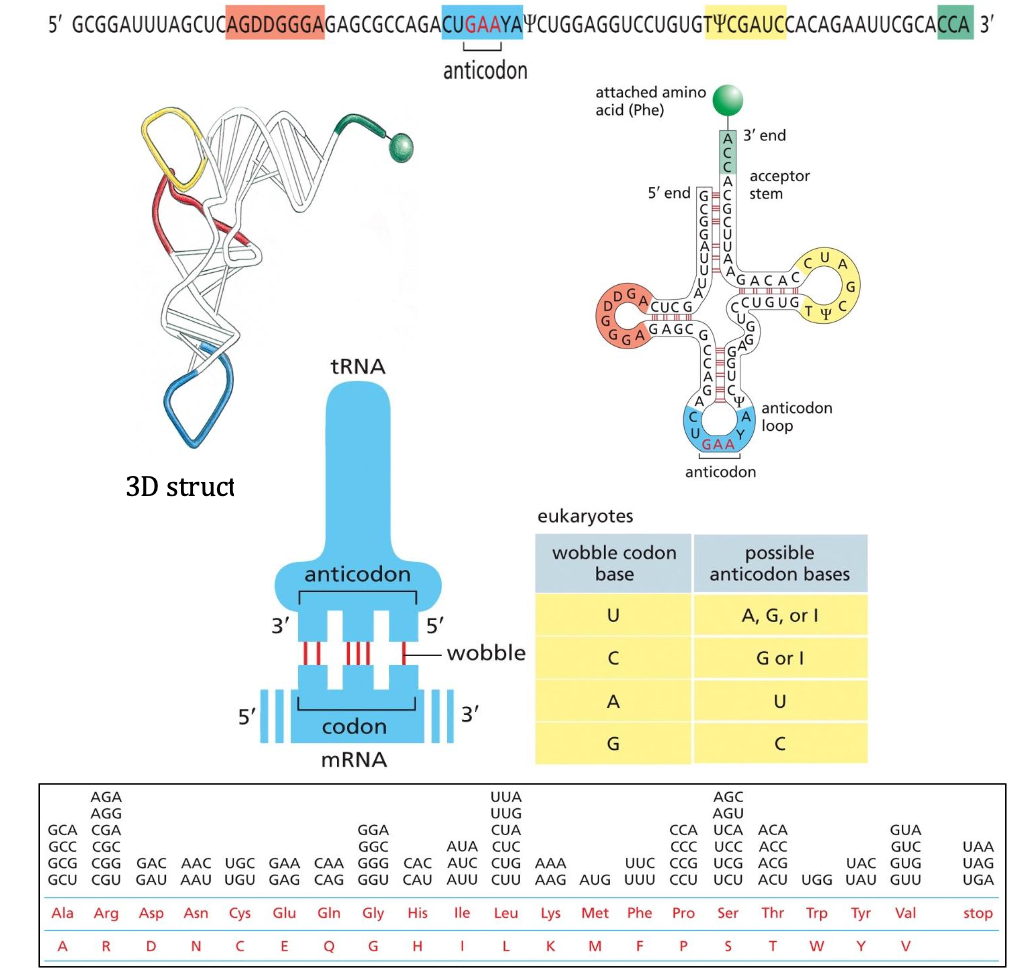
בתמונה האנזים הכתום מצמיד ל-tRNA את חומצת האמינו המתאימה.
מזהה גם את האנטי קודון וגם את קצה 3’ של ה-tRNA.
מצמידה רק לאחר זיהוי קומפלמנטרי נכון.
מחבר את החומצה אמינית ל-tRNA באמצעות קשר אסטרי.
יצירת הקישור
האנזים מקשר כימית בין חומצת האמינו ל-tRNA תוך שימוש ב-ATP, ויוצר קשר אסטרי יציב אך עדין.
איכות הזיהוי ומנגנוני בקרה
דיוק הזיהוי הכפול מבטיח נאמנות תרגום גבוהה (שגיאה של פחות מ-1 ל-10,000). חלק מהאנזימים כוללים גם אתר “editing” המסוגל לתקן שגיאות ולהסיר חומצות אמינו שגויות.
Wobble Base Pairing והקוד הגנטי - זיווג רופף
מנגנון המאפשר גמישות בקוד הגנטי תוך שמירה על דייקנות:
חוקי ה-Wobble
- עמדות 1 ו-2: חייב להיות זיווג מושלם 100%
- עמדה 3: מותר זיווג “רופף” לפי כללים ספציפיים:
- A יכול להתזווג רק עם U
- U יכול להתזווג עם A, G, או I (inosine)
- G יכול להתזווג עם C או U
- C יכול להתזווג רק עם G
אין כאן T - RNA של tRNA, אלא רק של mRNA.
משמעויות הביולוגיות:
- הסבר לכך שמספר קודונים מקודד לחומצת אמינו אחת
- קיומם של פחות מ-61 tRNAs שונים (למרות 61 קודונים מקודדי חלבון)
- גמישות אבולוציונית בקוד הגנטי
- הקטנת ההשפעה של מוטציות “שקטות”
תהליך התרגום המפורט
התחלת התרגום ותפקיד גורמי העזר
זיהוי ה-mRNA ותפקיד ה-Cap
כזכור, רק ב-mRNA יש Caping ו-Zn tail.
- תת-היחידה הקטנה (40S) מזהה את ה-7-methylguanosine cap (m^7G) בקצה 5’ של ה-mRNA
- גיוס translation initiation factors רבים (eIF1, eIF1A, eIF4F complex ועוד)
- יצירת קומפלקס סריקה הכולל 40S + eIFs + Met-tRNA
- סריקה לאורך ה-5’ UTR במהירות של כ-4 נוקלאוטידים/שנייה
היחידה ממשיכה עד שהיא מוצאת את ה-start codon (AUG) הראשון.
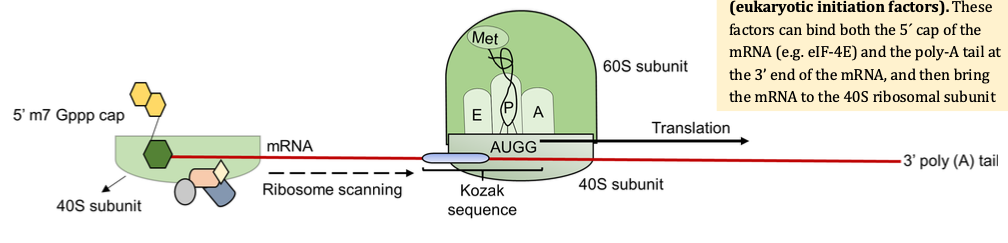
Kozak Sequence וזיהוי Start Codon
רצף הקונסנסוס: 5’-GCCRCCAUGG-3’ (כאשר R = פורין)
- המוטיב שמור אבולוציונית ונקרא על שם מרלין קוזק שגילתה אותו
- מבטיח התחלת תרגום במקום הנכון ומונע “התחלות כוזבות”
- באיקריוטים: Kozak sequence
- בפרוקריוטים: Shine-Dalgarno sequence (רצף שונה לחלוטין)
גיוס תת-היחידה הגדולה
לאחר זיהוי האתר הנכון:
- גיוס תת-היחידה הגדולה (60S)
- שחרור גורמי ההתחלה
- יצירת ריבוזום פעיל (80S) מוכן לתרגום
מחזור התרגום והאתרים הפעילים
הריבוזום מכיל שלושה אתרים מרכזיים המתפקדים בצורה מתואמת:
- A site (aminoacyl site): כניסת tRNAs טעונים חדשים
- P site (peptidyl site): מיקום ה-tRNA הנושא את השרשרת הגדלה
- E site (exit site): יציאת tRNAs ריקים
שלבי המחזור המפורטים
- Binding ובדיקה:
- tRNA טעון מגיע עם elongation factor Tu (eEF1A)
- בדיקת התאמה קודון-אנטי-קודון
- אם התאמה נכונה: קישור יציב; אם לא: דחייה מהירה
- Translocation:
- peptidyl transferase (פעילות קטליטית של ה-rRNA, לא חלבון!) יוצרת קשר פפטידי
- elongation factor G (eEF2) גורם להזזת הריבוזום 3 נוקלאוטידים
- tRNA עובר מ-A ל-P, מ-P ל-E
- שחרור: tRNA ריק יוצא דרך E site
מהירות התהליך:
- איקריוטים: 3-5 חומצות אמינו/שנייה
- פרוקריוטים: 20-15 חומצות אמינו/שנייה (מהיר יותר בגלל פשטות מערכתם)
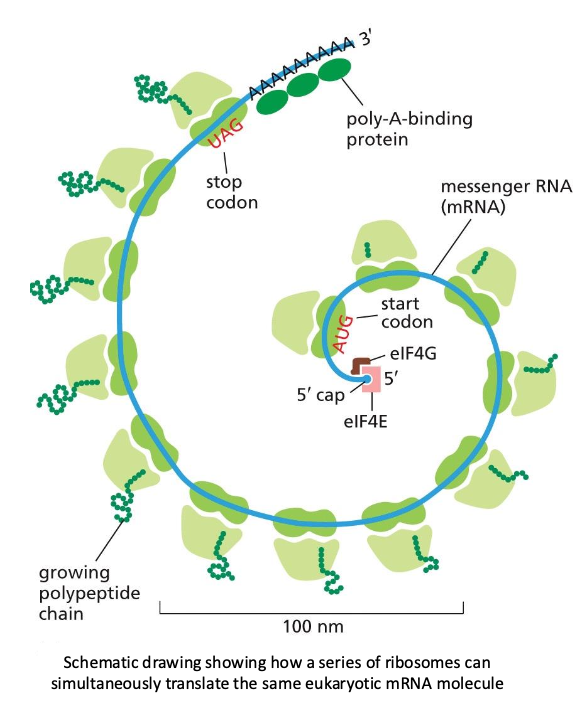
פוליריבוזומים ויעילות התרגום
מנגנון המאפשר תרגום יעיל של mRNA אחד על ידי מספר ריבוזומים במקביל:
התהליך:
- ריבוזומים מתחילים תרגום בזמנים מדורגים
- mRNA ארוך יכול לשאת 20-10 ריבוזומים או יותר
- כל ריבוזום מייצר עותק שלם של החלבון
- הריבוזום הראשון שהתחיל כמעט סיים, האחרון רק מתחיל
יתרונות:
- ייצור מקבילי של עותקים רבים
- ניצול מיטבי של ה-mRNA
- תגובה מהירה לצרכי התא
פוליריבוזומים קשורי-ER
ריבוזומים חופשיים מול קשורים
- ריבוזומים חופשיים: מייצרים חלבונים לציטופלזמה
- ריבוזומים קשורי-ER: מייצרים חלבונים להפרשה או לממברנות
מנגנון הקישור:
רצף אות (signal sequence) בתחילת החלבון הנוצר מזוהה על ידי signal recognition particle (SRP), המכוון את הריבוזום ל-ER.
סיום התרגום
התרגום מסתיים כאשר הריבוזום פוגש אחד משלושת stop codons:
- UAG (amber)
- UAA (ochre)
- UGA (opal)
במקום tRNA, מגיעים release factors (eRF1 ו-eRF3) המובילים לשחרור החלבון ופירוק קומפלקס התרגום.
השוואה בין פרוקריוטים לאיקריוטים
הבדלים מבניים ותפקודיים
מהירות תהליכים
| תהליך | איקריוטים (יצורים בעלי גרעין) | פרוקריוטים (בעיקר חיידקים) |
|---|---|---|
| שעתוק | 30-10 נוקלאוטידים לשנייה | מהיר יותר |
| תרגום | 5-3 חומצות אמינו לשנייה | 20-15 לשנייה |
הפרדה מרחבית
באיקריוטים קיימת הפרדה גרעין-ציטופלזמה המאפשרת:
- עיבוד מורכב של ה-RNA לפני התרגום
- ויסות נוסף ברמת הטרנספורט
- מניעת תרגום של mRNAs לא בשלים
- שליטה עדינה יותר על הביטוי הגנטי
מורכבות המנגנונים:
איקריוטים מפתחים מנגנונים מורכבים יותר (alternative splicing, מודיפיקציות רבות) שמאפשרים גיוון גדול יותר למרות מספר גנים דומה.
מודיפיקציות פוסט-טרנסלציוניות - מבוא
לאחר שהחלבון משוחרר מהריבוזום, הוא אינו מוכן עדיין לתפקוד. החלבון חייב לעבור מודיפיקציות נוספות הכרחיות לתפקודו התקין, המכונות מודיפיקציות פוסט-טרנסלציוניות (co-translational ו-post-translational modifications).
סוגי המודיפיקציות העיקריים
- גליקוזילציה: הוספת קבוצות סוכר ב-ER ובגולגי
- פוספורילציה: הוספת קבוצות פוספט לויסות פעילות
- אצטילציה: מודיפיקציה המשפיעת על פעילות ויציבות
- יוביקוויטינציה: סימון לפירוק חלבונים
- חיתוך: הסרת רצפי אות או אזורים לא נחוצים
מודיפיקציות אלו מתרחשות בעיקר ב-endoplasmic reticulum, במערכת הגולגי ובאברונים ספציפיים אחרים. נושא זה יכוסה בפירוט רב בשיעורים העוקבים.
סיכום וחשיבות הנושא
תהליכי עיבוד ה-RNA ותרגום החלבונים מהווים את הבסיס לכל התפקודים התאיים. הבנת מנגנונים אלו חיונית להבנת:
- מחלות גנטיות רבות
- פיתוח תרופות
- הנדסה גנטית
- מחקר בסיסי בביולוגיה תאית
המורכבות העצומה של מנגנונים אלו מעידה על ההתפתחות האבולוציונית הארוכה והעדינה שהביאה ליצירת מכונות מולקולריות מדויקות ויעילות המאפשרות את החיים כפי שאנחנו מכירים אותם.